近几年,国内外重大动物源性食品、生物制品安全事件频发。2013 年初,欧盟“马肉风波”[1,2]、2018年上旬出现的“虹鳟三文鱼”事件[3,4]、2018 年10 月24 日,中华人民共和国科技部公布处罚信息,多家国内著名医院和生物公司违反了人类遗传资源管理规定[5],将人类遗传资源违规运送出境。海关总署随后立即开展了进出境动物源性生物材料监管相关工作。这一领域的分子生物学检测技术基础研究和方法的建立工作亟需开展。
动物源性制品的掺假、标签误标、瞒标等是关系到人类健康和社会发展的重要问题。物种鉴定方法是非常重要的研究领域。由于具有高度遗传稳定性和种间遗传信息特异性,线粒体DNA(mtDNA)基因如细胞色素b[6-8],12S和16S[9],细胞色素C氧化酶I(COI)[10,11],D-loop[12]等区域经常被选择作为靶基因区域用于物种鉴定。目前,最常用的物种鉴定方法多基于聚合酶链式反应(Polymerasechain reaction,PCR)。如常规PCR 法[13],实时荧光PCR 技术(Real- time fluorescence polymerase chain reaction,qPCR)[14,15],环介导等温扩增技术(Loop-mediated isothermal amplification method,LAMP)[16,17],液滴式数字PCR法(droplet digital PCR technology,ddPCR)[18-20]。然而,上述检测方法均存在一定技术瓶颈,即它们都只能针对一种已知物种信息的物种进行有针对性的检测。如果待测样品是混合样品或完全无法预知的物种,则这些方法均不适用。
二代测序技术(The Next Generation Sequencing technology,NGS)[21]和DNA 条形码技术(DNA barcode)[22-24]以其自身的技术优势迅速成为全球分类学、生态学、进化学、遗传学等领域的研究热点。本研究中,摆脱传统的物种鉴定的思路(使用特异性的引物探针对某个物种进行一对一检测),综合利用这两项新技术的优势,根据各物种线粒体基因寻找两端具有足够保守性而中间序列具有高区分度的区域进行全新的通用型引物设计,通过大量比对和验证来确保该通用引物兼顾核酸扩增所需的保守性以及物种区分的特异性。再结合二代测序的高通量能力以及后续生物信息学分析,可以在一次测序反应中获得来自于混合样本中所有目标片段的序列,然后将这些序列与相应的数据库进行比对即可确定其代表物种。从而初步实现了对复杂背景样本和未知物种样本中各种动物源性成分信息的高通量检测。
1 材料与方法
1.1 材料与试剂
表1实验中所使用的动物源性标准品信息
Table 1Information on animal - derived standard substances used in the experiments

序号 物种名称 拉丁文 1牛 Bos taurus 2 绵羊 Ovis aries 3山羊Capra hircus 4 猪 Sus scrofa 5鸡 Gallus gallus 6 鸭 Anas platyrhynchos 7 鹅 Anser anser 8火鸡 Meleagris gallopavo 9 鹌鹑Coturnix coturnix 10 鸽子Columba livia 11驴Equus asinus 12 马Equus caballus 13水牛 Bubalus bubalis 14 猫Felis catus 15大西洋三文鱼 Salmo salar 16 虹鳟 Oncorhynchus mykiss 17 鳗鱼 Anguilla japonica 18鸵鸟Struthio camelus 19 袋鼠 Macropus rufus 20 梅花鹿 Cervus nippon 21骆驼 Camelus bactrianus 22 东北虎 Panthera tigris altaica 23黑冠长臂猿 Nomascus gabriellae 24 狮子Panthera leo
本实验中所使用的动物源性标准品信息见表1。所有动物源性标准品均为实验室保存材料。1-18 号物种标准品购自于Zyagen Laboratories(美国)公司;19-24号野生动物物种标准品由上海动物园提供。经一代测序鉴定并确认物种信息准确性和代表性。在上海本地超市和电商平台购置了10 个市售商业化样本,如猪肉火腿肠、清真鸡肉火腿肠,风干骆驼肉等,见表6。核酸提取试剂为美国OMEGA 公司,M9145 Mag-Bind® Viral DNA/RNA 96 Kit(50 μL);核酸纯化使用普洛麦格(北京)生物技术有限公司,Wizard Genomic DNA Purification kit;普通PCR 使用宝生物工程(大连)有限公司Premix Taq™(Ex Taq™ Version 2.0);核酸定量使用美国通用医疗GE NanoVue Plus超微量分光光度计和赛默飞世尔科技(中国)有限公 司Qubit dsDNA BR Assay Kit 核酸定量试剂盒;Illumina Miseq 高通量测序平台相关测序试剂等。
1.2 仪器与设备
VeritiTM 96梯度PCR 仪,美国 Applied Biosystems公司;Nanovue Plus微量分光光度计,英国GE 公司;Invitrogen Qubit® 2.0 荧光定量仪,赛默飞世尔科技(中国)有限公司;Life ViiA™ 7 实时荧光定量PCR仪,美国Applied Biosystems 公司;Illumina Miseq 高通量测序仪,美国illumina 公司。
1.3 方法
1.3.1 引物设计
首先根据动物线粒体全长基因寻找两端序列具有足够保守而中间序列具有高种间区分度的区域进行通用型引物设计并进行后续实验优化。根据后续二代测序平台的特点,PCR 产物的长度控制在350 bp 左右,这样既保证了足够的物种特异性信息量,也确保靶基因长度符合NGS 平台片段读取长度限制。与此同时,通过筛选试验进一步确保该引物对不同物种核酸具有基本相似的扩增效率。本研究重新设计、合成、优化、筛选了该通用引物对。在理论上,利用NCBI在线生物信息学软件工具(http://blast.ncbi.nlm.nih.gov/Blast.cgi)进行序列搜索和比对。结果表明,该引物能有效扩增常见哺乳动物、鸟类和鱼类的核酸。引物由生工生物工程(上海)股份有限公司合成。
表2通用引物序列
Table 2 Primer sequences for universal amplification experiment
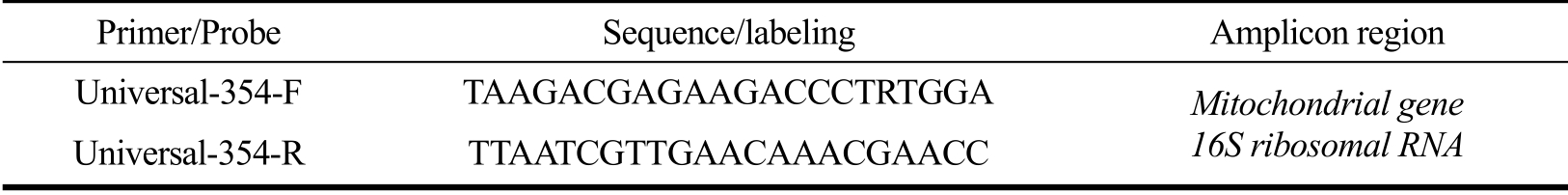
Primer/Probe Sequence/labeling Amplicon region Universal-354-F TAAGACGAGAAGACCCTRTGGA Mitochondrial gene 16S ribosomal RNA Universal-354-R TTAATCGTTGAACAAACGAACC
1.3.2 核酸提取
使用磁珠法DNA 提取试剂盒对实验中所有的动物源性样品进行核酸提取,具体操作步骤见试剂盒说明书。使用Nanovue Plus 微量分光光度计对DNA 样品的浓度纯度进行检测。每个样品同时检测3次取平均值,并用无菌水将质量浓度调整至4 ng/μL,-20 ℃储存备用。
1.3.3 PCR 扩增
使用Premix Taq 酶对所有动物源性样品核酸进行PCR 扩增。PCR 扩增体系:TaKaRa Ex10×Ex Taq Buffer(Mg2+ Plus)5μL,dNTP Mixture (各2.5 nM)4 μL,上游引物和下游引物(终浓度1μM)各0.5μL TaKaRa Ex Taq 酶(5U/μL)0.25μL,模板DNA 5μL,加灭菌蒸馏水补齐至50 μL。PCR 扩增程序:95 ℃预变性10 s;98 ℃变性10 s,55℃退火30 s,72 ℃延伸30s,35 个循环;72 ℃再延伸2 min;4℃保存。PCR 产物。每个物种的PCR 扩增产物使用琼脂糖凝胶电泳检测。各取2 μL 的PCR 产物于1.5%的琼脂糖凝胶(含溴化乙锭)中,电压120 V,电泳45min,在凝胶成像系统上拍照并分析引物的扩增效果。
1.3.4 sanger 测序
为了保证实验中所有涉及的动物源性核酸物种特异性信息的准确,利用一代测序技术,即Sanger 测序法对每个单一物种的核酸进行了测序比对。进行测序所有特定物种的准确性,每一个单一物种的DNA 样本都采用Sanger 测序法进行测序。首先,根据制造商的说明,使用AmpliTaq Gold™360 Master Mix(Applied Biosystems,Foster City,USA,California,USA)扩增靶基因模板。PCR 产物采用ExoSAP-IT(Affymetrix Inc,Santa Clara,CA,USA)处理样本。然后,使用BigDye Terminator v3.1Cycle Sequencing Kit(Thermo Fisher Scientific Corporation,Carlsbad,CA,USA)进行扩增。扩增产物经过纯化步骤后,装入ABI3730 毛细管测序仪(美国加州福斯特市应用生物系统公司)进行测序。最后使用Chroma(Technelysium Pty Ltd,South Brisbane,Australia)和Bioedit(http://www.mbio.ncsu.edu/BioEdit)对产生的电图进行分析和可视化检查。获得的序列使用在线生物信息学比对软件 blast(https://blast.ncbi.nlm.nih.gov/Blast.cgi)进行物种分析鉴定以识别每个样本的代表性物种。
1.3.5 二代测序
采用Agencourt AMPure® XP Reagent(Beckman Coulter)对扩增产物中的游离引物和引物二聚体进行纯化。引入不同的barcode 用于区分不同来源的样品。每个文库的浓度测定均使用Qubit®2.0Green 双链DNA 法。安捷伦公司的Bioanalyzer®2100和Agilent DNA 1000试剂盒被用于文库质量控制。根据测序覆盖率的需要,所有已标记barcode的文库都应该被汇集起来用于一次测序。最后使用美国Illumina 公司MiSeq 系统按照制造商的说明和步骤进行测序操作。建库和测序均委托生工生物工程(上海)股份有限公司完成。
1.3.6 数据分析
根据重叠片段信息,利用PEAR(v0.9.6)软件进行Illumina 短片段组装。然后对fastq 文件进行处理,生成相应的fasta 和qual 文件,便于在后续的标准化数据比较分析过程中使用。使用cutadapt 软件(v1.2.1,https://pypi.org/project/cutadapt/1.2.1/)和Prinseq 软件(v0.20.4, http://prinseq.sourceforge.net/)修剪长度小于200 bp 或大于400 bp 的序列,去除含有歧义碱基的序列。利用UCHIME(v4.2.40,http://drive5.com/usearch/ manual/uchime_algo.html)、Usearch(version 5.2.236,http://www.drive5.com/usearch)和BLASTN 去除扩增过程中产生的嵌合序列。最后使用Usearch(version 5.2.236,http://www.drive5.com/usearch/)将所有干净的数据聚类到Operational Taxonomic Units(OTU),相似性97%。使用BLASTN 将每个OTU 中丰度最高的序列与数据库进行比对。序列的相似性和覆盖率均满足>95%的,用于后续分类。不满足条件或有效比例小于0.5%的序列认定为未检出标记为未分类。
1.3.7 模拟样本和商业化样本测序分析
动物制品来源广泛,加工工艺多样,成分复杂。为验证该方法在实际动物源性产品中的准确性和适用性,在实验室内人工制作了已知成分的模拟样品并收集了商场中销售的10 种商业化产品进行了测序分析。
2 结果与分析
2.1 引物设计与扩增
表3不同物种靶序列区域上下游引物比对结果
Table 3Comparison results of upstream and downstream primers in target sequences of different species
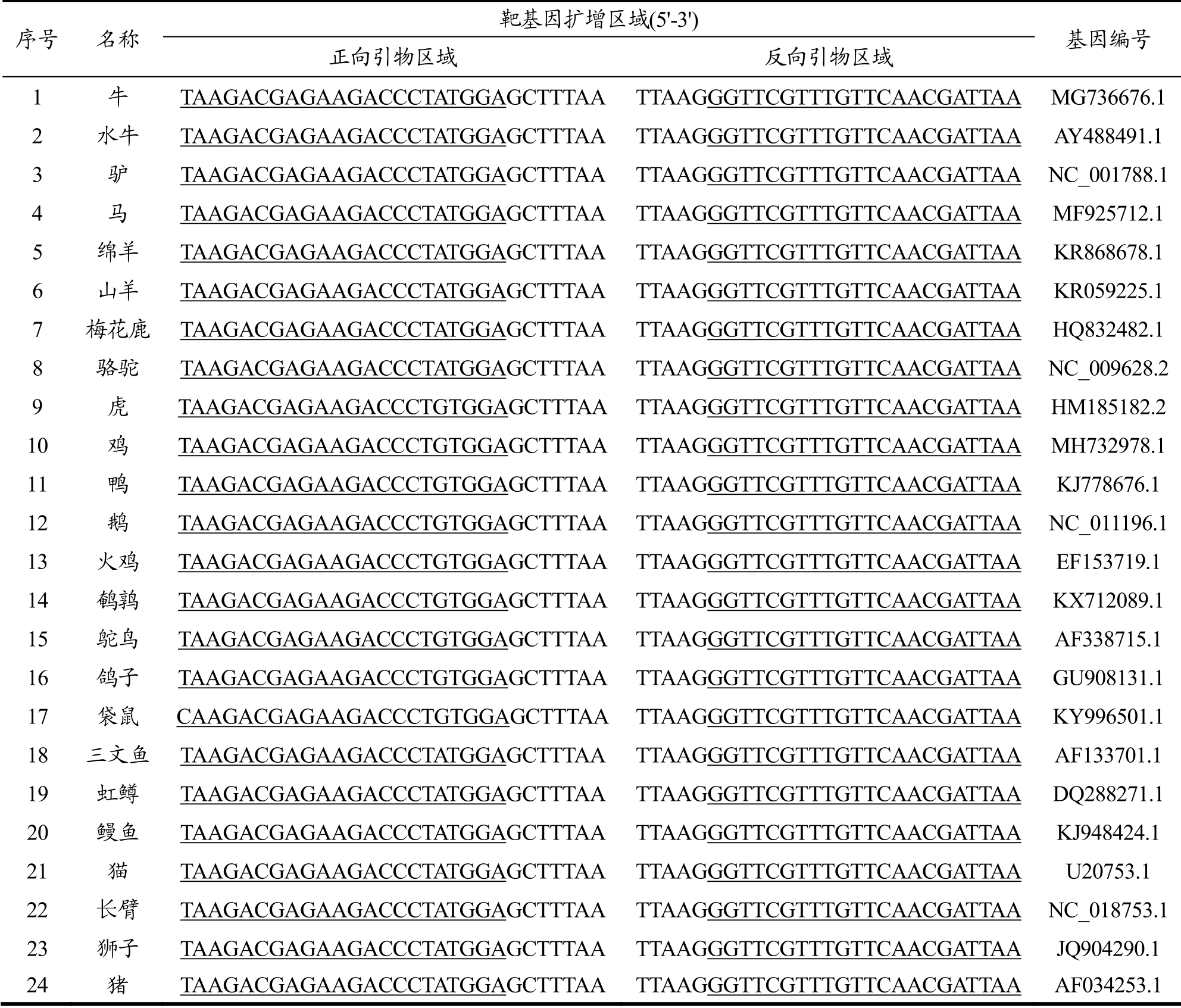
注:表中带下划线区域为通用引物结合区域。
序号 名称 靶基因扩增区域(5'-3') 基因编号正向引物区域 反向引物区域 1牛 TAAGACGAGAAGACCCTATGGAGCTTTAA TTAAGGGTTCGTTTGTTCAACGATTAA M 2水牛 TAAGACGAGAAGACCCTATGGAGCTTTAA TTAAGGGTTCGTTTGTTCAACGATTAA A 3驴 TAAGACGAGAAGACCCTATGGAGCTTTAA TTAAGGGTTCGTTTGTTCAACGATTAA NC 4马TAAGACGAGAAGACCCTATGGAGCTTTAA TTAAGGGTTCGTTTGTTCAACGATTAA M 5绵羊 TAAGACGAGAAGACCCTATGGAGCTTTAA TTAAGGGTTCGTTTGTTCAACGATTAA K 6 山羊 TAAGACGAGAAGACCCTATGGAGCTTTAA TTAAGGGTTCGTTTGTTCAACGATTAA K 7 梅花鹿 TAAGACGAGAAGACCCTATGGAGCTTTAA TTAAGGGTTCGTTTGTTCAACGATTAA H 8骆驼 TAAGACGAGAAGACCCTATGGAGCTTTAA TTAAGGGTTCGTTTGTTCAACGATTAA NC 9虎 TAAGACGAGAAGACCCTGTGGAGCTTTAA TTAAGGGTTCGTTTGTTCAACGATTAA H 10鸡 TAAGACGAGAAGACCCTGTGGAGCTTTAA TTAAGGGTTCGTTTGTTCAACGATTAA M 11鸭 TAAGACGAGAAGACCCTGTGGAGCTTTAA TTAAGGGTTCGTTTGTTCAACGATTAA K 12鹅 TAAGACGAGAAGACCCTGTGGAGCTTTAA TTAAGGGTTCGTTTGTTCAACGATTAA NC 13火鸡 TAAGACGAGAAGACCCTGTGGAGCTTTAA TTAAGGGTTCGTTTGTTCAACGATTAA E 14 鹌鹑 TAAGACGAGAAGACCCTGTGGAGCTTTAA TTAAGGGTTCGTTTGTTCAACGATTAA K 15鸵鸟TAAGACGAGAAGACCCTGTGGAGCTTTAA TTAAGGGTTCGTTTGTTCAACGATTAA A 16 鸽子 TAAGACGAGAAGACCCTGTGGAGCTTTAA TTAAGGGTTCGTTTGTTCAACGATTAA G 17 袋鼠 CAAGACGAGAAGACCCTGTGGAGCTTTAA TTAAGGGTTCGTTTGTTCAACGATTAA K 18三文鱼 TAAGACGAGAAGACCCTATGGAGCTTTAA TTAAGGGTTCGTTTGTTCAACGATTAA A 19 虹鳟 TAAGACGAGAAGACCCTATGGAGCTTTAA TTAAGGGTTCGTTTGTTCAACGATTAA D 20 鳗鱼 TAAGACGAGAAGACCCTATGGAGCTTTAA TTAAGGGTTCGTTTGTTCAACGATTAA K 21猫TAAGACGAGAAGACCCTATGGAGCTTTAA TTAAGGGTTCGTTTGTTCAACGATTAA 22长臂TAAGACGAGAAGACCCTATGGAGCTTTAA TTAAGGGTTCGTTTGTTCAACGATTAA NC 23狮子 TAAGACGAGAAGACCCTATGGAGCTTTAA TTAAGGGTTCGTTTGTTCAACGATTAA JQ 24猪 TAAGACGAGAAGACCCTATGGAGCTTTAA TTAAGGGTTCGTTTGTTCAACGATTAA A G736676.1 Y488491.1_001788.1 F925712.1 R868678.1 R059225.1 Q832482.1_009628.2 M185182.2 H732978.1 J778676.1_011196.1 F153719.1 X712089.1 F338715.1 U908131.1 Y996501.1 F133701.1 Q288271.1 J948424.1 U20753.1_018753.1 904290.1 F034253.1
在理论上,利用在线生物信息学分析软件(http://blast.ncbi.nlm.nih.gov/Blast.cgi)进行序列搜索和比对。结果表明,该引物能有效扩增常见哺乳动物、鸟类和鱼类的核酸。扩增出的不同物种的序列位置如表3所示。另一方面,为了验证引物在实际应用中的适用性,对实验室保存的哺乳动物、鸟类、鱼类和植物等常见物种特异性DNA 样本进行了扩增。琼脂糖电泳结果显示,所有样品均获得了明亮单一的扩增条带,相似的灰度表明各物种核酸扩增效率基本相似,未见非特异性扩增(图1)。实验结果说明设计的通用型引物对不同动物基因组中的靶基因具有较好的适用性。与已经报道的通用引物[25]相比较,本研究中设计优化的引物在保证了高效扩增效率和较好适用范围的前提下,为后续测序片段比对工作提供了更为丰富的序列信息,有助于提高物种鉴定的分辨率。

图11-24 号物种特异性核酸PCR扩增产物琼脂糖电泳结果
Fig.1 Agarose electrophoresis results of 1-24 species-specific nucleic acid PCR amplification products
2.2 sanger 测序:鉴定物种特异性序列
对本实验中所有涉及的动植物样本DNA 使用通用引物(Universal-354-F,Universal-354-R)分别进行扩增,所得扩增产物均需要经sanger 测序认定物种信息。测序片段均使用Blast 中的Nucleotide collection(nr/nt)数据库默认设置进行比对。24个物种DNA测序结果均与数据库中比对出来的代表性物种一致。实验结果表明,该通用引物对这24 个哺乳动物、禽类和鱼类样本核酸具有较广的适用范围和较高的扩增效率。图1 各个物种扩增产物条带亮度相似,也说明该通用引物对不同物种核酸的扩增效率接近。确定了物种信息的DNA 样品用于后续的二代测序实验。所有DNA 样本均保存于-80 ℃冰箱。
2.3 混合DNA 样品检测分析
为了验证该测序鉴定方法的准确性,实验室内人工配制了2组不同物种特异性DNA 的混合样本。Sample1:十二种动物(牛、驴、绵羊、山羊、猪、马、骆驼、鸡、鸭、鹅、鹌鹑和猫)DNA 混合比例相同。每个物种的最终浓度约8.3 ng/μL。Sample 2:12 种动物(牛、驴、绵羊、山羊、猪、马、骆驼、鸡、鸭、鹅、鹌鹑和猫)和5 种植物(小麦、高粱、豌豆、燕麦、土豆)DNA 混合比例相同。每个物种的最终浓度约5.9ng/μL。通过扩增建库、测序和数据比对分析,实验结果清楚地显示了混合样品中每个预添加物种的准确信息。从结果中可以看出,即使是多种不同动物和植物物种基因组DNA 混合物(图2a、b),该检测方法仍然可以清晰地显示出每种成分的特异性信息。检测结果没有物种间干扰现象出现。测序结果中没有发现漏检或假阳性现象。植物基因组DNA 无法检测到,且对其他动物物种的鉴定没有影响(图2b)。实验验证了该方法的准确性。混合DNA样本sample 1总reads数目为75160,测序深度为72187×;sample 2 总reads数目为222778,测序深度为213967×。

图2已知成分配比混合DNA 样品测序检测鉴定结果
Fig.2Sequencing and identification results of mixed DNA samples known as distribution ratios
2.4 模拟动物源性样品检测
动物制品来源广泛,加工工艺多样,成分复杂。为了验证该方法在实际动物源性产品中的适用性,在实验室内按照肉丸制作方法制作了两个模拟肉丸样本。其中肉丸1 使用不同质量绞碎的山羊肉、马肉、火鸡肉、牛肉、猪肉;肉丸2 使用等质量绞碎的鹅肉、马肉和小麦粉、玉米粉、大豆粉。添加适量食盐、味精、酱油、胡椒粉等佐料搅拌混匀并捏制成丸后煮熟。随后对两个模拟样品进行采样并进行测序检测分析。实验结果显示,模拟样品1 和2 中预添加的所有动物源性成分均可以在一次测序检测中检测到。模拟样本肉丸2 中小麦、大豆、玉米三个植物源性成分则无法检测到(表4)。没有发现误检或遗漏。也没有发现日常食品中使用的佐料或者加热这样的烹饪方式对检出结果有影响。这一结果证实了该二代测序检测方法的准确性和实用性。
表4 已知成分的模拟样本测序检测结果
Table 4 Sequencing test results of simulated samples with known components
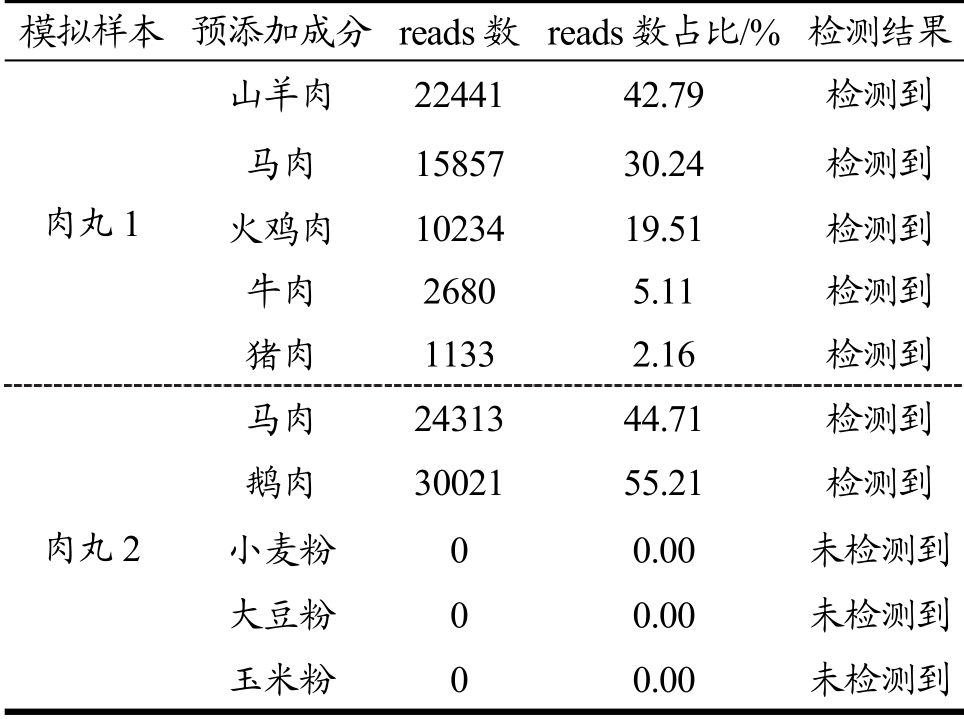
模拟样本 预添加成分 reads 数 reads 数占比/% 检测结果 肉丸1山羊肉2244142.79 检测到马肉15857 30.24 检测到火鸡肉10234 19.51检测到牛肉2680 5.11检测到猪肉11332.16 检测到肉丸2 马肉2431344.71检测到鹅肉3002155.21检测到小麦粉 0 0.00 未检测到大豆粉 0 0.00 未检测到玉米粉 0 0.00 未检测到
2.5 已知浓度占比动物源性样品检测
表5已知浓度占比混合样本三次测序检测结果
Table 5 Sequencing test results of mixed samples with known concentration ratio
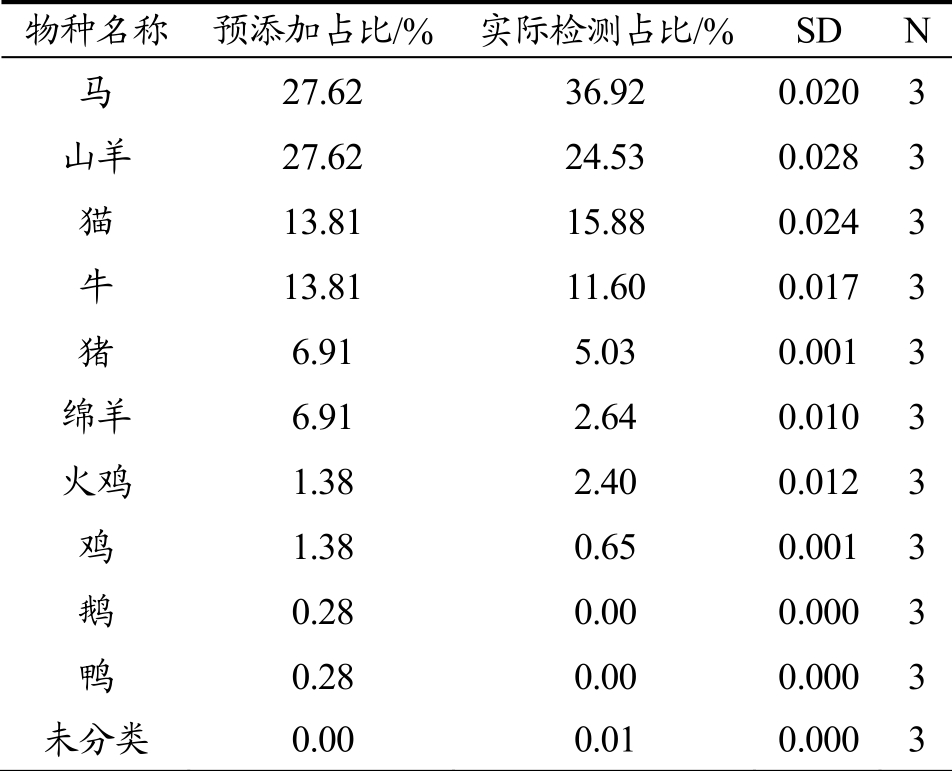
物种名称 预添加占比/% 实际检测占比/% SD N 马27.62 36.92 0.020 3山羊27.62 24.53 0.028 3猫13.81 15.88 0.024 3牛13.81 11.60 0.017 3猪 6.91 5.03 0.001 3绵羊6.91 2.64 0.010 3火鸡 1.38 2.40 0.012 3鸡1.38 0.65 0.001 3鹅0.28 0.00 0.000 3鸭0.28 0.00 0.000 3未分类0.00 0.01 0.000 3
不同物种基因组DNA 按已知比例进行混合。原液浓度分别为马、山羊(100 ng/μL),猫、牛(50 ng/μL),猪、绵羊(25ng/μL),火鸡、鸡(5 ng/μL),鹅、鸭(1 ng/μL)。每个物种DNA 各取10μL,则混合液中10 个物种DNA 终浓度分别为马、山羊(10 ng/μL),猫、牛(5 ng/μL),猪、绵羊(2.5 ng/μL),火鸡、鸡(0.5ng/μL),鹅、鸭(0.1 ng/μL)。混合液中各物种间DNA 比例为100:50:25:5:1。充分混匀后单独进行三次测序检测鉴定实验。实验结果显示(表5),山羊、猫、猪、火鸡的预计占比分别为27.62%、13.81%、6.91%和1.38%,而三次平行检测结果平均值分别为24.53%、15.88%、5.03%和2.40%。各物种检测结果与预添加比例具有相对一致性。说明该方法不但能够区混合样本中物种成分,而且具备对每个组分含量进行初步定量检测的潜力。但实验中发现,该引物对不同物种靶基因扩增效率存在一定差异,这也导致同一浓度组的两个物种的实际检测占比与预添加占比产生一定差异,说明如果需要进行定量检测,则后续还需要对引物的扩增效率进一步优化。在1ng/μL浓度水平的鹅、鸭两个预添加物种均未能检出,也证明了该二代测序物种鉴定方法存在最低检测下限。实验结果显示,当鹅和鸭的核酸在初始混合液中浓度为0.5 ng/μL时,本测序方法无法得到有效的特异性物种信息。
2.6 商业化样本测序分析
表6商业化样本测序检测结果
Table 6 Sequencing test results of commercial samples

商品名称 标签成分 检测成分 猪肉火腿肠猪 猪和鸡 猪肉馅猪 猪 清真鸡肉火腿肠鸡 鸡风干鹿肉鹿 鹿 羊肉罐头羊羊牛肉干牛 马和牛 鸡肉米饭狗粮 鸡 鸡酱驴肉驴驴风干骆驼肉骆驼 骆驼 酱板鸭 鸭 鸭
商业化动物源性制品种类繁多,样本特性不相同。为了验证该测序检测方法在面对不同特性样本的适应性,该测序方法被用来对市售样本进行了完整流程的检测实验。测试结果显示,10 个样品中有2 个不符合标签。检测结果表明,猪肉火腿肠中发现标签中未标识的鸡核酸成分;牛肉干中也发现了马成分(表6),这些实验结果证明了该NGS检测方法具有广泛的适用范围和较高的实用性。通过对10 种市售商业化动物源性制品的采集、前处理、核酸提取、测序分析,全流程检测后发现,实验涉及的所有的样品都可以用这种方法完成测序检测。虽然对于一些脂肪含量高的样品,核酸提取效率会降低,但在后续的核酸扩增测序过程中没有发现无法完成检测的现象。
3 结论
本研究针对生物制品中动物源性成分鉴定的短板和存在的问题,提出、建立并优化了一种基于大规模通用靶基因平行扩增测序策略。首次使用全新设计和优化的通用型引物。通过序列比对和PCR 扩增实验,证明该引物能够以相似的扩增效率有效扩增多种哺乳动物,禽类和鱼类的核酸。扩增产物的长度接近350 bp,具有较高的物种特异性分辨率。通过进一步的实验,验证了该方法的准确性,灵敏度和检测下限。特别是在针对市售的10个商业化动物源性制品样本测序检测后发现了两例标签不符问题。这不单说明市售商品确实存在问题,还证明了该检测方法的实际应用能力。此外,该物种鉴定系统具有较好的开放性,不仅适用于哺乳动物,禽类和鱼类,而且可以通过选择适当的引物组和生物信息学分析方法应用于昆虫和植物物种的鉴定。
[1]O'Mahony,P J.Finding horse meat in beef products--a global problem [J].QJM, 2013,106(6):595-7
[2]Nau,J Y.Horse meat:first lessons of a scandal [J].Rev Med Suisse,2013, 9(376):532-3
[3]朱梓明.从虹鳟被列为三文鱼看团体标准[J].质量与标准化,2018(10):28-30 ZHU Zi-ming.Rainbow trout is listed as salmon group standard [J].Quality and standardization, 2018, 10:28-30
[4]丁清龙,曾晓琮,周露,等.三文鱼水产品掺假情况调查[J].食品安全质量检测学报,2019,10(13):4080-4085 DING Qing-long,ZENG Xiao-cong,ZHOU Lu,et al.Investigation of salmon adulteration [J].Journal of Food Safety and Quality.2019,10(13):4080-4085
[5]韩静.贯彻落实《中华人民共和国人类遗传资源管理条例》中华医学会倡导进一步加强医学伦理规范建设[J].中华医学信息导报,2019.34(13):2 HAN Jing.Implementation of the regulations of the People's Republic of China on the management of human genetic resources,Chinese medical association advocates further strengthening the construction of medical ethics [J].China Medical News, 2019, 34(13):2
[6]Rasmussen R S,Morrissey M T.DNA-based methods for the identification of commercial fish and seafood species[J].Comprehensive Reviews in Food Science and Food Safety,2008,7(3): 280-295
[7]李宗梦,赵良娟,马兴,等.肉及肉制品中大鼠成分PCR 检测方法研究[J].食品研究与开发,2016,37(24):109-113 LI Zong-meng,ZHAO Liang-juan,MA Xing,et al.Study on PCR detection of rat components in meat and meat products [J].Food Research and Development, 2016, 37(24):109-113
[8]吕明星,唐俊妮,陈娟,等.用Cytb基因序列分析鉴定肉制品掺假[J].中国食品卫生杂志,2016,28(1): 48-51 LYU Ming-Xing,TANG Jun-ni, CHEN Juan, et al.Identification of meat products adulteration using Cytb gene sequence analysis [J].Chinese Journal of Food Hygiene,2016, 28(1):48-51
[9]周彤,李家鹏,李金春,等.一种基于多重实时荧光聚合酶链式反应熔解曲线分析的肉及肉制品掺假鉴别方法[J].食品科学,2017,38(12):217-222 ZHOU Tong,LI Jian-peng, LI Jin-chun,et al.A method for identification of adulteration of meat and meat products based on multiple real-time fluorescence polymerase chain reaction melting curve analysis [J].Food Science,2017,38(12):217-222
[10]刘建辉,唐先明,杜丽丽,等.羊猪鸭基因组DNA 提取及DNA 条形码分子鉴定[J].哈尔滨商业大学学报(自然科学版),2018,34(6):.658-662 LIU Jian-hui,TANG Xian-ming,et al.Genomic DNA extraction and DNA barcode molecular identification of sheep and ducks [J].Journal of Harbin University of Commerce (Natural Science Edition),2018,34(6):658-662
[11]王爽,李永波,马超峰,等.DNA 条形码COI 序列在常见肉类鉴别中的应用研究[J].现代食品科技,2016,32(1):188-193 WANG Shuang,LI Yong-Bo,MA Chao-Feng,et al.Application of DNA barcode COI sequence in common meat identification [J].Modern Food Science and Technology,2016,32(1):188-193
[12]章晶晶,杜利强,李永艳,等.实时荧光PCR 法快速鉴别狐狸貉子肉源性成分研究[J].现代食品科技,2017,33(8):269-275 ZHANG Jing-jing,DU Li-qiang,LI Yong-yan,et al.Rapid identification of meat-derived components of fox scorpion by real-time fluorescent PCR [J].Modern Food Science and Technology,2017,33(8): 269-275
[13]刘国强,海小,罗建兴,等.PCR 技术鉴定肉品中鸭源性成分的方法研究 [J].中国家禽,2018,40(24):58-60 LIU Guo-qiang,HAI Xiao,LUO Jian-xing,et al.A method for the identification of duck-derived components in meat by PCR [J].Chinese poultry,2018, 40(24):58-60
[14]Kim M,Yoo I,Lee SY,et al.Quantitative detection of pork in commercial meat productsby TaqMan(R)real-time PCR assay targeting the mitochondrial D-loop region [J].Food Chem, 2016, 210: 102-106
[15] Fang, X, C Zhang. Detection of adulterated murine components in meat products by TaqMan(c) real-time PCR [J]. Food Chem, 2016. 192: 485-490
[16] 杨柳,张一,王磊,等.环介导等温扩增技术在羊肉制品掺假检测中的应用[J].黑龙江畜牧兽医(上半月),2017,3:272- 273 YANG Liu, ZHANG Yi, WANG Lei, et al. Application of loop-mediated isothermal amplification technique in adulteration detection of mutton products [J]. Heilongjiang Animal Husbandry and Veterinary Medicine (First Half of the Month), 2017, 3: 272-273
[17] 朱凯,康怀彬,王德国,等.可视化LAMP 检测常见肉制品中猪肉成分[J].食品科学,2019,40(12):296-302 ZHU Kai, KANG Huai-bin, WANG De-guo, et al. Visualization of LAMP for the detection of pork in common meat products [J]. Food Science, 2019, 40(12): 296-302
[18] Cai Y, He Y, Lv R., et al., Detection and quantification of beef and pork materials in meat products by duplex droplet digital PCR [J]. PLoS One, 2017, 12(8): e0181949
[19] Cai Y, Li X, Lv R, et al., Quantitative analysis of pork and chicken products by droplet digital PCR [J]. Biomed Res Int, 2014, 2014: 810209
[20] Bertrand C, Janzen D H, Hallwachs W, et al., Mitochondrial and nuclear phylogenetic analysis with sanger and next-generation sequencing shows that, in area de conservacion guanacaste, northwestern costa rica, the skipper butterfly named Urbanus belli (family hesperiidae) comprises three morphologically cryptic species [J]. BMC Evol Biol, 2014, 14: 153
[21] Zhou X G, Ren L F, Li Y T, et al. The next-generation sequencing technology: a technology review and future perspective [J]. Sci China Life Sci, 2010, 53(1): 44-57
[22] Ståhlberg A, Krzyzanowski P M, Egyud M, et al. Simple multiplexed PCR-based barcoding of DNA for ultrasensitive mutation detection by next-generation sequencing [J]. Nat Protoc, 2017, 12(4): 664-682
[23] Sarwat M, M M Yamdagni. DNA barcoding, microarrays and next generation sequencing: recent tools for genetic diversity estimation and authentication of medicinal plants [J]. Crit Rev Biotechnol, 2016. 36(2): 191-203
[24] Shokralla S, Gibson J F, Nikbakht H, et al. Next-generation DNA barcoding: using next-generation sequencing to enhance and accelerate DNA barcode capture from single specimens [J]. Mol Ecol Resour, 2014, 14(5): 892-901
[25] Bertolini F, Ghionda M C, D’Alessandro E, et al. A next generation semiconductor based sequencing approach for the identification of meat species in DNA mixtures [J]. PLoS One, 2015, 10(4): e0121701