图1 16S rDNA扩增片段图
Fig.1 Agarose gel electrophoresis of 16S rDNA PCR products
摘要:细菌是雪莲菌中的重要功能菌群,对于雪莲菌的形成及其功能作用的发挥有着重要作用,因而探明其细菌的种群特征具有重大意义。本文采用聚合酶链式反应-变性梯度凝胶电泳(PCR-DGGE)和16S rDNA测序技术对3个西藏雪莲菌颗粒(A、B和C)中细菌多样性进行了研究。DGGE电泳结果显示,样品的丰度值介于12~16之间,细菌多样性指数在1.79~2.11之间,相似性指数在0.62~0.77之间。通过对DGGE指纹图谱优势条带切胶测序结果显示:它们分别属于马乳酒样乳杆菌(Lactobacillus kefiranofaciens)、乳球菌属(Lactococcus)、乳杆菌属(Lactobacillus)、嗜热链球菌(Streptococcus thermophilus)、开菲尔乳杆菌(Lactobacillus kefir)、气单胞菌属(Aeromonas)和醋酸菌属(Acetobacter)。旨在为雪莲菌微生物资源开发以及加快雪莲菌工业化生产奠定基础。
关键词:雪莲菌;聚合酶链式反应;变性梯度凝胶电泳;细菌多样性
雪莲菌又名西藏灵菇,呈乳白色、胶质状的块状物,外形酷似小块花椰菜[1],经过长期在牛奶中培养后,个体逐渐增大,犹如盛开的雪莲,是一种传统酒精发酵乳饮料的发酵剂。经雪莲菌发酵后的发酵乳,是我国西藏和新疆等地居民的传统食品,除了具有酸奶特有的酸味和香味以外,还具有轻微的醇香及起泡性,更重要的是它还有抑菌活性、抗氧化活性、改善肠道提高免疫力、抗肿瘤活性和抗炎症活性等保健功效[2,3],因而备受人们关注。然而,雪莲菌发酵乳只能通过雪莲菌颗粒进行生产,这种传代方式不稳定性从而限制了其工业化生产,并且目前人工条件下还不能合成雪莲菌颗粒,而关于其成粒机理至今也无法解释。雪莲菌与开菲尔粒类似,目前普遍认为两者为发源于不同地方的同一种物质,国外对开菲尔粒的研究起步较早,一些国家已经开始使用从开菲尔粒中分离的不同菌种制备纯培养发酵剂[4],用于开发不同种类的功能性酸奶产品,因此对雪莲菌颗粒中微生物资源研究,对于开发我国丰富的微生物资源以及拓宽乳制品工业发展新思路具有重要意义。
雪莲菌中的微生物由乳酸菌和酵母菌等组成,它们赋予了雪莲菌发酵乳特殊的风味和价值。大量研究采用传统分离方法对雪莲菌的微生物组成进行了分离鉴定,但微生物传统分离方法复杂繁琐,往往不能获得微生物组成的全部信息。近年来,随着分子生物学技术不断完善和发展,越来越多的免培养技术逐渐被应用于雪莲菌中微生物的研究。聚合酶链式反应(polymerase chain reaction,PCR)配合变性梯度凝胶电泳(denaturing gradient gel electrophoresis,DGGE)技术通过直接提取样品DNA,能有效分析微生物群落多样性,可以充分还原菌落结构,是研究发酵食品中微生物多样性的有力手段。周剑忠等[5]、林晓珊等[6]和许女等[7]采用 PCR-DGGE技术对雪莲菌中的微生物组成进行研究,对其优势种群结构特征有了一定认识,但样品单一、对于不同来源样品的比较以及分析优势菌群,微生物多样性的研究相对较少。本研究采用PCR-DGGE以及16S rDNA测序技术对来自西藏的3个不同雪莲菌中细菌多样性进行分析,经DNA提取、巢式PCR、DGGE电泳和克隆测序后,对优势菌株进行了分离鉴定,分析优势菌群,探讨细菌多样性,为雪莲菌中细菌多样性研究以及微生物资源的开发奠定基础。
1.1 样品
雪莲菌:自西藏不同地区采集3个雪莲菌颗粒,放于10%(m/V)的无菌牛奶中,置于28 ℃的环境中,每隔24 h更换1次牛奶,连续培养5 d。
1.2 试剂与仪器
Tris、TEMED、尿素、醋酸、过硫酸铵、丙烯酰胺、甲叉双丙烯酰胺、苯酚、氯仿和SDS,阿拉丁试剂(上海)有限公司;DNA Marker、琼脂糖、溶菌酶、EB核酸染料、Proteinase K(10 mg/mL)、RNase(10 mg/mL)、TaKaRa Taq、10×PCR buffer、dNTP Mixture 和pMD-18-T载体宝生物工程大连有限公司。所有引物合成及测序由北京鼎国昌盛生物技术有限责任公司完成。
梯度 PCR仪、VerSa Doc凝胶成像系统和DCodeTM System 变性梯度凝胶电泳系统,美国Bio-Rad公司。
1.3 方法
1.3.1 雪莲菌基因组提取以及V3区PCR
参照Nalbantoglu[8]中的方法,在无菌条件下对雪莲菌颗粒充分研磨,采用苯酚-氯仿抽提的方法对雪莲菌基因组进行提取,然后琼脂糖凝胶电泳检测后,置于-4 ℃保存以备用。
以提取的雪莲菌基因组 DNA为模板,用 16S rDNA通用引物27f/1492r进行扩增,扩增产物长度分别约为1500 bp。扩增体系为DNA模版:1 μL,dd水:37 μL,10×PCR Buffer:5 μL,dNTP:4 μL,正反向引物:各1 μL,rTaq酶:1 μL。反应程序:95 ℃预变性5 min,94 ℃变性1 min,58 ℃退火40 s,72 ℃延伸90 s,30个循环,72 °延伸10 min。扩增产物经0.8%琼脂糖凝胶电泳验证后,作为巢式PCR的模版。
以 16S rDNA PCR产物为模版,采用通用引物338f-gc-clamp/518r对16S rDNA的V3区进行扩增,扩增产物长度分别约为250 bp。扩增体系以及反应程序与以上相同。扩增产物经 2%琼脂糖凝胶电泳检测后,置于-20 ℃冰箱保存备用,作为后续DGGE分析的样品。
1.3.2 变性梯度凝胶电泳
采用美国Bio-Rad公司的DcodeTM通用突变检测系统对PCR扩增产物进行电泳分离分析。分别取样品的巢式PCR扩增产物Loading Buffer混合后加入到点样孔中,采用 8%聚丙烯酰胺变性凝胶,变性剂梯度范围为30%~60%,在1×TAE缓冲液中,恒温60 ℃、恒压80 V条件下电泳720 min。电泳结束后取出胶放入200 mL EB染液中浸染10~20 min,使用紫外凝胶成像系统拍照。
1.3.3 DGGE指纹图谱分析
根据Quantity One软件读取DGGE图谱上不同条带的光密度值,进行match比对后进行聚类分析,生成 DGGE图谱相似性分析图。本研究主要使用丰度(S)、shannon-wiener多样性指数(H)和样品间相似性指数(Cs)来表示群落多样性。其计算公式如下:
(1)丰度(S):即DGGE图谱中条带的个数。
(2)Shannon-Wiener多样性指数[9]:即反映群落种类与均匀度的混合参数;同样,种类之间个体分配的均匀性增加也会使多样性提高。H=-Σpiln(pi)式中,H为Shannon-Wiener多样性指数;pi为第i个条带的优势度;S为每一个泳道的丰度值。
(3)相似性系数(Sorenson Pairwise Similarity Coefficient):通过利用Quantity one软件的进行聚类分析,采用UPGMA算法对三株雪莲菌样品进行分析。
1.3.4 DGGE片段的回收、克隆与测序
对目的条带切胶回收放入超纯水4 ℃过夜,取2 μL上清液,以不带GC夹子的细菌V3区可变区通用引物DNA片段进行扩增,反应体系及程序与巢式PCR一致。目标片段经 2%琼脂糖凝胶电泳检测后,置于-20 ℃冰箱保存备用,作为后续克隆的目的片段。采用pMD18-T Vector Cloning Kit对扩增片段进行克隆,将目的片段与大肠杆菌DH5α感受态细胞悬液过夜连接,进行蓝白斑筛选分别挑3个阳性克隆子至10 mL LB液体培养基中,37 ℃、180 r/min培养8 h后用试剂盒提取质粒。以提取质粒DNA为模板,使用M13F 和M13R引物进行扩增,扩增产物用2%琼脂糖凝胶电泳检测后,送北京金唯智公司测序。
1.3.5 系统发育学分析
对测序所得的序列进行去除载体片段,得到目标序列。登陆NCBI,将所得序列与数据库中已知序列进行比较,得到各克隆子的种属信息。根据比对结果可以知道,克隆子在进化上的亲缘关系,用Clustal X 和DNAStar进行相似性分析。
2.1 雪莲菌细菌16S rDNA V3区扩增结果

图1 16S rDNA扩增片段图
Fig.1 Agarose gel electrophoresis of 16S rDNA PCR products
图2 16S rDNA V3区扩增片段图
Fig.2 Agarose gel electrophoresis of 16S rDNA V3 region
以提取的3个雪莲菌基因组DNA为模板,首先采用27f/1492r引物对3个雪莲菌A、B和C的基因组DNA进行16S rDNA特异性扩增,扩增产物长度分别约为1500 bp左右(图1)。再以其PCR产物为模版,使用338f-gc-clamp/518r引物进行16S rDNA V3区PCR扩增,得到250 bp左右的片段(图2),符合引物设计片段。
2.2 DGGE电泳结果及指纹图谱丰度及优势度比较
采用 8%聚丙烯酰胺变性凝胶,变性剂梯度范围为30%~60%,对细菌16S rDNA V3区进行DGGE电泳,结果显示:3个样品分离得到12~16条清晰的条带,表明所有样品的16S rDNA V3片段通过DGGE电泳得到了很好的分离,结果如图3所示:
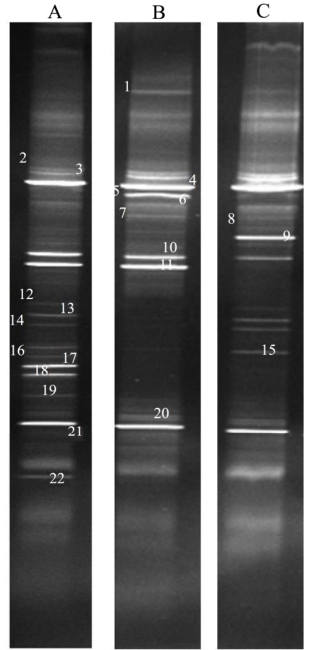
图3 16S rDNA V3区PCR-DGGE指纹图谱
Fig.3 PCR-DGGE analysis of 16S rDNA V3 region
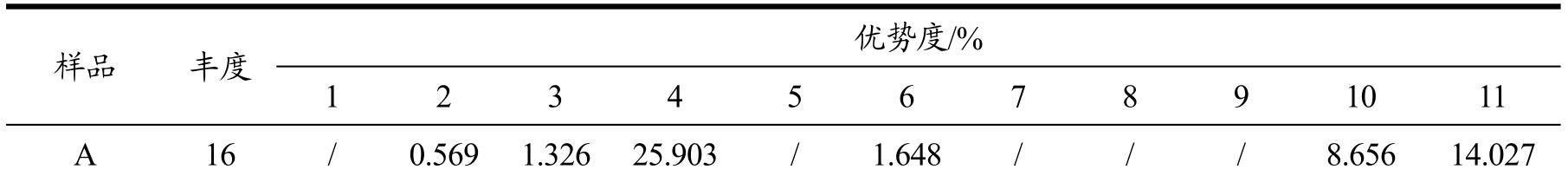
本研究分析了3个雪莲菌的细菌微生物群落的丰度以及优势度,通过DGGE指纹图谱可以看出,图谱中各条带都有相应的迁移率和光强度。从迁移率可以看出,样品之间具有很多相同的条带;3个雪莲菌的丰度在12~16之间,其中雪莲菌A的丰度最高,雪莲菌C次之,雪莲菌B最少为12;指纹图谱中5条条带在所有样品中均有检出,其中编号4和21优势度较高在7%以上,而编号10在雪莲菌A和B中均存在,含量也在4%以上;而条带5和9分别在雪莲菌B和C中检出,且含量在 5%以上,表明这些条带所代表的菌株可能在雪莲菌中起着重要的作用(表1)。
表1 雪莲菌细菌DGGE图谱的丰度和优势度
Table 1 Abundance and dominance of DGGE bands of bacteria in Tibetan kefir grains
?
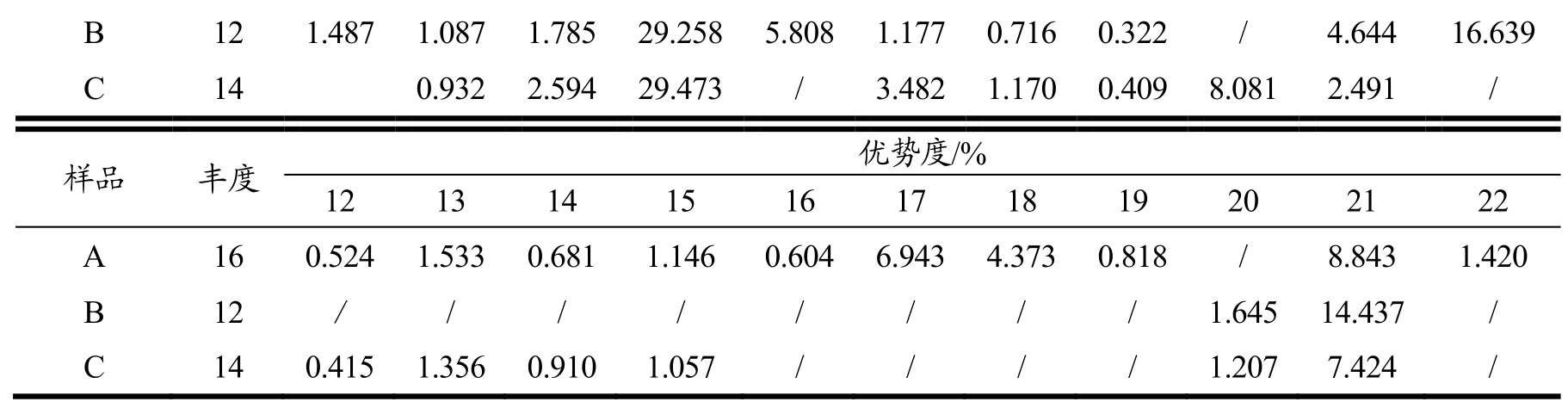
注:“/”为未检出。
B 12 1.487 1.087 1.785 29.258 5.808 1.177 0.716 0.322 / 4.644 16.639 C 14 0.932 2.594 29.473 / 3.482 1.170 0.409 8.081 2.491 /样品 丰度 优势度/% 12 13 14 15 16 17 18 19 20 21 22 A 16 0.524 1.533 0.681 1.146 0.604 6.943 4.373 0.818 / 8.843 1.420 B 12 / / / / / / / / 1.645 14.437 / C 14 0.415 1.356 0.910 1.057 / / / / 1.207 7.424 /
2.3 雪莲菌细菌 DGGE指纹图谱多样性指数以及相似性比较重要参数,广泛应用于生物多样性的评价和研究。本文利用Shannon-Wiener指数以及UPGMA算法分析了3个雪莲菌的细菌DGGE图谱的多样性指数以及相似性值,结果表明(表2和图4),雪莲菌细菌多样性指数均在1.78~2.11之间,其中雪莲菌A的多样性指数为2.11,雪莲菌B为1.78,而雪莲菌C为1.94,可以看出雪莲菌A和C在群落多样性上比较接近。相似性指数分析中,一般认为相似值高于0.60的群体具有较好的相似性,由图4看出,3个雪莲菌的细菌相似性指数都在0.6以上,3个雪莲菌的细菌微生物组成差异较小,但A和B处在同一分支上,相似程度更高。
表2 雪莲菌细菌群落相似性指数
Table 2 Similarity of bacterial community in Tibetan kefir grains
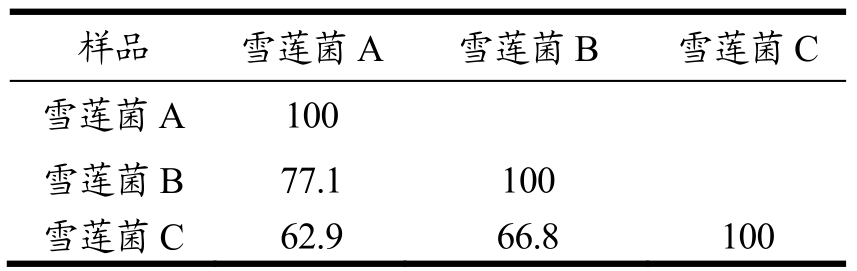
样品 雪莲菌A 雪莲菌B 雪莲菌C雪莲菌A 100雪莲菌B 77.1 100雪莲菌C 62.9 66.8 100
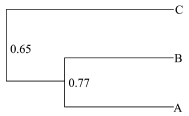
图4 不同雪莲菌细菌群落相似性指数树状图
Fig.4 Dendrogram with homology coefficient of different Tibetan kefir grains
微生物群落的多样性指数是微生物群落研究中的
2.4 雪莲菌细菌 DGGE图谱优势菌的测序分析
选取8条优势条带进行切胶回收测序,分别为雪莲菌B的4号、5号、10号和11号,雪莲菌C的9号和21号,以及雪莲菌A的17号和18号。测序结果经NCBI上BLAST比对结果见表3:
表3 DGGE指纹图谱条带序列比对结果
Table 3 Closest match identification of DGGE bands

菌株编号 BLAST结果 序列结果/bp 登录号 最大鉴定百分率/% B4 马乳酒样乳杆菌(Lactobacillus kefiranofaciens) 198 KT368993.1 100 B5 马乳酒样乳杆菌(Lactobacillus kefiranofaciens) 198 KT368992.1 100 C9 乳球菌属(Lactococcus) 198 KM021302.1 99 B10 乳杆菌属(Lactobacillus) 198 JQ394820.1 100 B11 嗜热链球菌(Streptococcus thermophiles) 199 KT124584.1 100 A17 开菲尔乳杆菌(Lactobacillus kefir) 198 KC845576.1 99 A18 气单胞菌属(Aeromonas) 198 KT727994.1 100 C21 醋酸菌属(Acetobacter ) 173 KP966444.1 100
2.5 雪莲菌 DGGE图谱优势条带系统发育学分析
8条优势条带在系统发育树上可以分为6个类群(图 5所示),它们分别属于马乳酒样乳杆菌( Lactobacillus kefiranofaciens)、 乳 球 菌 属(Lactococcus)、乳杆菌属(Lactobacillus)、嗜热链球菌(Streptococcus thermophilus)、开菲尔乳杆菌(Lactobacillus kefir)、气单胞菌属(Aeromonas)和醋酸菌属(Acetobacter)。
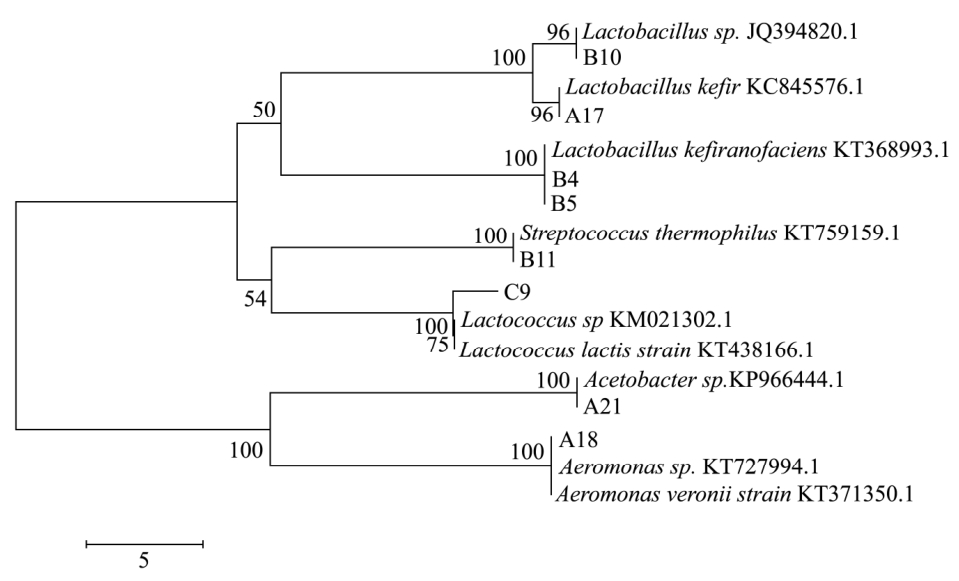
图5 DGGE条带序列与GenBank数据库中相关序列的相似性分析
Fig.5 Similarity analysis of the sequences derived from DGGE bands and related sequences from GenBank
马乳酒样乳杆菌(Lactobacillus kefiranofaciens)在3个雪莲菌中均存在,且含量最高,优势度在25%以上,表明其是3个雪莲菌中主要的、最易被检测出来的乳杆菌。Arihara K[10]利用免疫荧光显微镜来检测到马乳酒样乳杆菌分布于整个开菲尔颗粒中并向中心增加。而在雪莲菌B的DGGE指纹图谱中,B-1和B-2虽然处于不同的位置,但均为马乳酒样乳杆菌,这可能与 PCR-DGGE所使用的V3区引物有关。Magalhaes[11]指出通过使用16S rDNA V3区片段来进行PCR-DGGE,不能区分开一些种的乳酸菌,同时一种乳杆菌在DGGE指纹图谱中不止一条带,这可能由于16S rDNA多个拷贝之间的靶序列的异质性。
醋酸菌属(Acetobacter)也均存在于所有的雪莲菌样品中,优势度均在 7%以上,含量相对较高。而在一些研究结果表明醋酸菌并不是雪莲菌颗粒微生物中的必要组成部分,与其来源有关。Mu等人[12]指出当醋酸菌与链球菌同时存在的时候,能够提高最终的酸度,增强酸化能力,同时也可以增强产品的粘度从而提高稳定性,可以看出醋酸菌有着至关重要的作用,需要进一步试验验证。
乳球菌属(Lactococcus)、乳杆菌属(Lactobacillus)和嗜热链球菌(Streptococcus thermophilus)等菌群存在于3个雪莲菌中,这些菌株能产胞外多糖以及其他代谢产物,对雪莲菌的结构组成和它所特有的粘性和微观结构,以及雪莲菌发酵乳的风味和营养价值都至关重要。而在雪莲菌A中首次发现气单胞菌属,有研究[13]表明该菌具有致病力,关于它的来源与危害需进一步研究。由于雪莲菌菌相比较复杂,其微生物组成与来源和培养条件有关,而一些研究表明雪莲菌或者开菲尔粒中或多或少的存在着致病菌[14]。因此,对雪莲菌的微生物组成进行快速有效的研究,加快纯培养发酵剂的开发具有重要意义。
雪莲菌作为天然的传统发酵剂,不同地区,不同来源的雪莲菌制作的发酵乳也存在一定的差异,而且人们至今也无法在不使用原粒的情况下合成新粒,因此对雪莲菌中微生物组成的研究具有重大意义。本实验采用PCR-DGGE和16S rDNA测序技术对来自西藏的3个不同雪莲菌中细菌多样性进行分析,与前人的研究相比,样品的丰度值和细菌多样性更为丰富,相似性指数在0.6以上,表明这3个雪莲菌的细菌多样性差异较小,相似性较高。DGGE指纹图谱中优势条带测序显示乳球菌属、乳杆菌属、链球菌属和醋酸菌属为主要优势菌,表明这些菌种对雪莲菌颗粒的发酵性能、功能特性以及成粒特性的贡献具有重要作用。同时我们确立了马乳酒样乳杆菌和醋酸菌属为3个样品中的主要优势菌,优势度分别在25%和7%以上,关于其作用值得进一步研究,而这种混合菌相的组合发酵模式为发酵乳行业乃至整个食品行业拓宽了思路,丰富发酵乳制品的市场。本文分析了来自西藏地区的3个雪莲菌颗粒的细菌多样性,对雪莲菌中的真菌微生物组成还可以进一步研究。与此同时该研究也提示我们,对于新疆和青海等其他地区的雪莲菌还可以进行系统研究,分析优势菌群,探讨微生物多样性,为雪莲菌微生物资源的开发以及加快雪莲菌工业化生产奠定基础。
参考文献:
[1] Marshall V M, Cole W M, Brooker B E. Observations on the structure of kefir grains and the distribution of the microflora [J]. Journal of Applied Microbiology, 1984, 57(3): 491-497
[2] 毛健,王豪,苏米亚,等.开菲尔中生物活性物质的益生作用[J].中国乳品工业,2010,38(12):30-35 MAO Jian, WANG Hao, SU Mi-ya, et al. Study on probiotic effect originated from bioactive ingredients in kefir [J]. China Dairy Industry, 2010, 38(12): 30-35
[3] 齐红霞.藏灵菇发酵乳的抗疲劳作用研究[D].长春:吉林大学,2008 QI Hong-xia. The study on the antifatigue effects of fermented milk of Zanglinggu [D]. Changchun: Jilin University, 2008
[4] Assadi M M, Pourahmad R, Moazami N. Use of isolated kefir starter cultures in kefir production [J]. World Journal of Microbiology and Biotechnology, 2000, 16(6): 541-543
[5] 周剑忠,董明盛,江汉湖.PCR-DGGE指纹技术与分离技术结合筛选藏灵菇奶发酵过程的优势菌[J].中国农业科学,2006,39(8):1632-1638 ZHOU Jian-zhong, DONG Ming-sheng, JIANG Han-hu. Screen of dominating microbial species isolated from tibetan kefir using an integrated approach of PCR-DGGE and culture-dependent methods [J]. Scientia Agricultura Sinica, 2006, 39(8): 1632-1638
[6] 林晓珊,阮征,刘飞,等.PCR-DGGE技术对中华开菲尔微生物菌群的分析[J].现代食品科技,2013,29(6):1217-1221 LIN Xiao-shan, RUAN Zheng, LIU Fei, et al. Analysis of microbial community structures in kefir grains by PCR-DGGE techniques [J]. Modern Food Science and Technology, 2013, 29(6): 1217-1221
[7] 许女,王艳萍,习傲登,等.西藏kefir粒中菌相的初步研究[J].中国酿造,2011,30(10):133-137 XU Nv, WANG Yan-ping, XI Ao-deng, et al. Mircobial population analysis of Tibet kefir [J]. China Brewing, 2011, 30(10): 133-137
[8] Nalbantoglu U, Cakar A, Dogan H, et al. Metagenomic analysis of the microbial community in kefir grains [J]. Food Microbiology, 2014, 41(4): 42-51
[9] Zoetendal E G, Akkermans A D, Vos W M D. Temperature gradient gel electrophoresis analysis of 16S rRNA from human fecal samples reveals stable and host-specific communities of active bacteria [J]. Applied & Environmental Microbiology, 1998, 64(10): 3854-3859
[10] Arihara K, Toba T, Adachi S. Immunofluorescence microscopic studies on distribution of Lactobacillus kefiranofaciens and Lactobacillus kefir in kefir grains [J]. International Journal of Food Microbiology, 1990, 11(2): 127-134
[11] Magalhães K T, Pereira G V M, Dias D R, et al. Microbial communities and chemical changes during fermentation of sugary Brazilian kefir [J]. World Journal of Microbiology and Biotechnology, 2010, 26(7): 1241-1250
[12] Mu Z, Yang X J, Yuan H. Detection and identification of wild yeast in koumiss [J]. Food Microbiology, 2012, 31(2): 301-308
[13] 王闻卿,苏靖华,傅慧琴,等.食源性气单胞菌属种水平监测和表型特征研究[J].中国食品卫生杂志,2014,26(1):1-5 WANG Wen-qing, SU Jing-hua, FU Hui-qin, et al. Monitoring and phenotype characteristics study on food-borne Aeromonas species [J]. Chinese Journal of Food Hygiene, 2014, 26(1): 1-5
[14] Chen H C, Wang S Y, Chen M J. Microbiological study of lactic acid bacteria in kefir grains by culture-dependent and culture-independent methods [J]. Food Microbiology, 2008, 25(3): 492-501
Analysis of the Bacterial Diversity in Tibetan Kefir Grains Using PCR-DGGE Techniques
Abstract:Bacteria are important functional microorganisms in Tibetan kefir grains, and play an important role in their formation and functions. Therefore, exploring the bacterial species in Tibetan kefir grains is of great significance. Polymerase chain reaction-denaturing gradient gel electrophoresis (PCR-DGGE) and 16S rDNA sequencing were used to study the bacterial diversity of three Tibetan kefir grains (A, B, and C) from different places. The DGGE results showed that the richness indices in Tibetan kefir grains were between 12 and 16, the diversity indices of all samples were between 1.79 and 2.11, and the similarity indices were between 0.62 and 0.77. The PCR-DGGE dominant bands were cut and sequenced, and the results indicated that the dominant bacteria belonged to Lactobacillus kefiranofaciens, Lactococcus spp., Lactobacillus spp., Streptococcus thermophilus, Lactobacillus kefir, Aeromonas spp., and Acetobacter spp. This work provides a theoretical basis for the development of microbial resources and the acceleration of the industrial production of Tibetan kefir grains.
Key words:Tibetan kefir grains; polymerase chain reaction; denaturing gradient gel electrophoresis; bacterial diversity
文章篇号:1673-9078(2017)2-83-88
DOI:10.13982/j.mfst.1673-9078.2017.2.013
收稿日期:2016-02-22
基金项目:国家863计划项目(2013AA102106)