食源性大肠杆菌O157毒力、耐药性及CRISPR分型分析
杨广珠,张淑红,黄远斌,吴清平,张菊梅
(华南应用微生物国家重点实验室,广东省菌种保藏与应用重点实验室,广东省微生物应用新技术公共实验室,广东省微生物研究所,广东广州 510070)
摘要:为深入了解食品源大肠杆菌O157的生物学特点,本研究对2014-2015年分离的39株大肠杆菌O157进行了PCR鉴定,毒力基因和耐药性检测,并利用规律成簇的间隔短回文重复序列(CRISPR)分型技术对菌株的遗传特性进行了分析。结果表明,39株菌株中有8株鉴定为O157:H7,31株为O157;检测的11种毒力基因中,eae存在于所有菌株中,stx2存在于82.50%菌株中,stx1未检出,其他毒力岛基因espA、etpD、tir、toxB、iha和katP携带率分别为92.31%、94.87%、87.18%、79.49%、69.23%和46.15%。药敏试验结果表明,菌株对四环素、复方新诺明、链霉素、氯霉素和氨苄西林等抗生素高度耐药,超过 30%菌株具有多重耐药性。CRISPR分型结果表明菌株具有较高的遗传多样性,39株菌株中有33株存在CRISPR1位点,8株E. coli O157:H7产生了相同的CRISPR spacer图谱,而26株E. coli O157产生了13种spacer图谱。本研究为食源性疾病的监测、疾病溯源和流行病学研究提供重要的基础数据。
关键词:大肠杆菌O157;毒力基因;耐药性;CRISPR分型
大肠杆菌Escherichia coli O157是一种重要的食源性致病菌,可引起人感染性腹泻、出血性结肠炎(hemorrhagic colitis,HC)和溶血性尿毒综合征(haemolytic-uraemic syndrome,HUS)等疾病,严重时可致人死亡[1,2]。近年来,国际上多个国家如美国、日本、英国等发生了O157食源性疾病暴发和流行,因此被列为重点监测致病菌之一[3]。我国已将大肠杆菌O157:H7列为21世纪对国人卫生健康有重大影响的12种病原微生物之一[4]。
Escherichia coli O157的致病性与多种毒力因子有关,已经公认的毒力因子有:志贺样毒素(Shiga-toxin,Stx)、LEE毒力岛和质粒 pO157[5,6]。志贺毒素是E. coli O157:H7最主要的毒力因子,包括Stx1和Stx2两种类型。Stx在引起腹泻、肠粘膜出血水肿、出血性肠炎和溶血性尿毒综合征等症状中起着重要作用。LEE毒力岛与细菌对粘附肠粘膜细胞和损伤相关。LEE毒力岛包含多个毒力基因:编码分泌型蛋白的espABD基因,编码紧密连接素的eae基因、编码紧密连接素受体的tir基因等。几乎所有E. coli O157:H7菌株含有一个约92 kb的大质粒(pO157),该质粒上的hly、katP、espP、toxB和stcE已被确认与细菌致病机制密切相关[6]。大肠杆菌O157:H7致病机理不是由单一毒力因子所决定,而是多种毒力因子共同作用的结果,监测毒力谱的变化非常重要。
细菌耐药性问题己经成为全球关注的一个问题。食源性致病菌耐药性的报道不断增多[7~9],给人类和动物健康带来极大的危害,监测这些细菌抗性谱的变化对临床选择抗生素具有重要指导意义。
近年来,多种分子分型技术被应用于食源性致病菌溯源和流行病学研究,如多位点序列分析(Multilocus sequence typing,MLST)、肠杆菌基因间共有重多序列多态性(Enterobacterial Repetitive Intergenic Consensus Polymerase Chain Reaction,ERIC-PCR)、随机扩增片段多态性分析(Random Amplified Polymorphic DNA,RAPD)等[10~12]。与传统分型技术相比,分子分型技术具有简便、快速和高分辨率等诸多优势。规律成簇的间隔短回文重复序列(Clustered Regularly Interspaced Short Palindromic Repeats,CRISPR)是一类重复结构,存在于 90%的古细菌和 40%的细菌基因组中,主要由重复序列(repeat)和间隔序列(spacer)等组成。一个细菌中可以有多个CRISPR位点,在每个CRISPR位点中重复序列几乎完全一致,而 spacer高度可变。CRISPR中的spacer对于不同菌株具有一定的识别意义,因此可用于致病菌遗传进化分析。作为一种新型分子分型方法,CRISPR已在沙门氏菌、大肠杆菌和金黄色葡萄球菌等致病菌分型中有所应用[13~15]。
目前国内系统完善的食源性致病菌信息数据库仍在不断更新中,本研究对2014~2015年全国食品样品中收集的 39株大肠杆 O157菌株进行了毒力因子检测、耐药性和CRISPR分子分型,旨在为食源性疾病的监测、溯源和流行病学研究提供重要的基础资料。
1 材料与方法
1.1 材料
1.1.1 菌株来源
本实验中39株性大肠杆菌O157菌株全部来源于2014~2015年全国污染调查的肉制品和蔬菜样品,其中来自牛肉12株,鸡肉20株,羊肉2株,猪肉2株和蔬菜 3株。所有菌株经过 API 20E生化鉴定条和O157单因子诊断血清鉴定为大肠杆菌O157。将菌株保藏在15%甘油管-80 ℃保存。参考标准菌株E. coli O157:H7 ATCC35150由广东省微生物分析检测中心惠赠。
1.1.2 主要试剂与仪器
细菌DNA提取试剂盒,购自北京天根生化科技有限公司;2×Taq PCR Master Mix、50×TAE、DNA Marker2000、琼脂糖,购自广州东盛生物科技有限公司;大肠杆菌O157显色培养基、胰蛋白胨大豆肉汤(TSB)、MH平板、营养琼脂平板,购自广东环凯微生物科技有限公司;药敏纸片,购自英国Oxiod公司;大肠杆菌O157和H7诊断血清,购自宁波天润生物药业有限公司;API 20E生化鉴定试剂条,购自广州千江企业有限公司。
Biometra TProfessional PCR仪(德国Biometra公司),电泳仪(EPS301、EPS501、EPS3501)(瑞典安玛西亚公司)和凝胶分析成像系统 UVI(英国 GE Healthcare公司)。
1.2 方法
1.2.1 双重PCR鉴定
将菌株接种到TSB肉汤,37 ℃培养24 h,取1 mL菌液于1.5 mL离心管中,12000 r/min离心5 min,弃上清。按细菌 DNA提取试剂盒说明书提取基因组DNA。利用本实验室建立的多重PCR方法,扩增O157抗原和H7抗原编码基因(rfbE和fliC)。引物序列和扩增片段大小见表 1,由华大基因合成。反应总体积为 25 μL,包括 2×PCR mix 12.5 μL,正反向引物各0.5 μL,模板 2 μL。以标准菌株 E. coli O157:H7 ATCC 35150为阳性对照。扩增条件:94 ℃预变性5 min;然后 94 ℃变性 30 s,58 ℃退火 30 s,72 ℃延伸 1 min。共进行30个循环,最后72 ℃延伸7 min。取8 μL PCR产物上在1.5%琼脂糖凝胶进行电泳,观察结果。
表1 本研究所用引物序列
Table 1 PCR primers used in this study

目的基因 引物序列(5′-3′) 片段大小/bp 参考文献eae GACCCGGCACAAGCATAAGC CCACCTGCAGCAACAAGAGG 384 bfpA AATGGTGCTTGCGCTTGCTGC GCCGCTTTATCCAACCTGGTA 326Paton and Paton,1998 stx1 ATAAATCGCCATTCGTTGACTAC AGAACGCCCACTGAGATCATC 180 stx2 GGCACTGTCTGAAACTGCTCC TCGCCAGTTATCTGACATTCTG 255 saa CGTGATGAACAGGCTATTGC ATGGACATGCCTGTGGCAAC 119 katP CTTCCTGTTCTGATTCTTCTGG AACTTATTTCTCGCATCATCC 2125 etpD CGTCAGGAGGATGTTCAG CGACTGCACCTGTTCCTGATTA 1062 toxB ATACCTACCTGCTCTGGATTGA TTCTTACCTGATCTGATGCAGC 602Monaghan et al.,2013 iha CAGTTCAGTTTCGCATTCACC GTATGGCTCTGATGCGATG 1305 tir CATTACCTTCACAAACCGAC CCCCGTTAATCCTCCCAT 1550 espA CACGTCTTGAGGAAGTTTGG CCGTTGTTAATGTGAGTGCG 299 espB CGATGGTTAATTCCGCTTCG GCCTGCTGAATCTGATAGCT 304 Our Lab fliCh7 GCGCTGTCGAGTTCTATCGAGC CAACGGTGACTTTATCGCCATTCC 625 rfbE TGTCCATTTATACGGACATCCATG CCTATAACGTCATGCCAA AT TGCC 291 Crispr1 GTTATGCGGATAATGCTACC CGTAYYCCGGTRGATTTGGA Y是C或T;R是A或G;M是A或C 1500 Liang,2016
1.2.2 毒力基因检测
根据参考文献合成毒力基因引物[16,17],引物序列及扩增片段大小见表1。PCR扩增反应体系:2×PCR mix 12.5 μL,浓度为 10 μmol/L 的上下游引物各 1 μL,DNA模板2 μL,补足灭菌双蒸水至25 μL。PCR扩增程序参考文献进行,以标准菌株 E. coli O157:H7 ATCC35150作为阳性对照,E. coli 25922和无菌水为阴性对照。
1.2.3 药敏实验
采用纸片琼脂扩散法(K-B法)进行药敏试验。将菌株在37 ℃营养肉汤中培养18~24 h,划线于营养琼脂平板表面,用生理盐水稀释至0.5麦氏浊度,涂布MH平板,然后用无菌镊子将药敏片贴于MH培养基表面,37 ℃培养24 h后测抑菌圈直径,根据美国临床实验室标准化协会(CLSI)标准判定菌株对药敏纸片的敏感性(表3)。所用16种药敏纸片包括:氨苄西林(AMP,10 μg)、阿莫西林/克拉维酸(AMC,30 μg)、头孢他啶(CAZ,30 μg)、头孢噻肟(CTX,30 μg)、头孢西丁(FOX,30 μg)、头孢噻吩(KF,30 μg)、环丙沙星(CIP,5 μg)、奈定酮酸(NA,30 μg)诺氟沙星(NOR,10 μg)、阿米卡星(AK,30 μg)、庆大霉素(CN,10 μg)、卡那霉素(K,30 μg)、链霉素(S,25 μg)、氯霉素(C,30 μg)、复方新诺明(SXT,25 μg)和四环素(TE,30 μg)。以大肠杆菌E. coli 25922为质控菌株。
1.2.4 CRISPR分子分型
按试剂盒法提取细菌基因组 DNA。参考文献[15]中的方法对菌株进行CRISPR1位点检测,CRISPR引物序列如表1所示,引物由华大基因合成。将CRISPR1的PCR扩增产物经2.0%琼脂糖凝胶电泳后,送华大基因公司测序将菌株的 CRISPR序列通过 CRISPR finder在线软件(http://crispr.u-psud.fr/Server/)进行分析并获取DR重复序列和spacer信息,并与数据库中的序列进行比对,绘制spacer排列图谱。
2 结果与分析
2.1 双重PCR鉴定结果
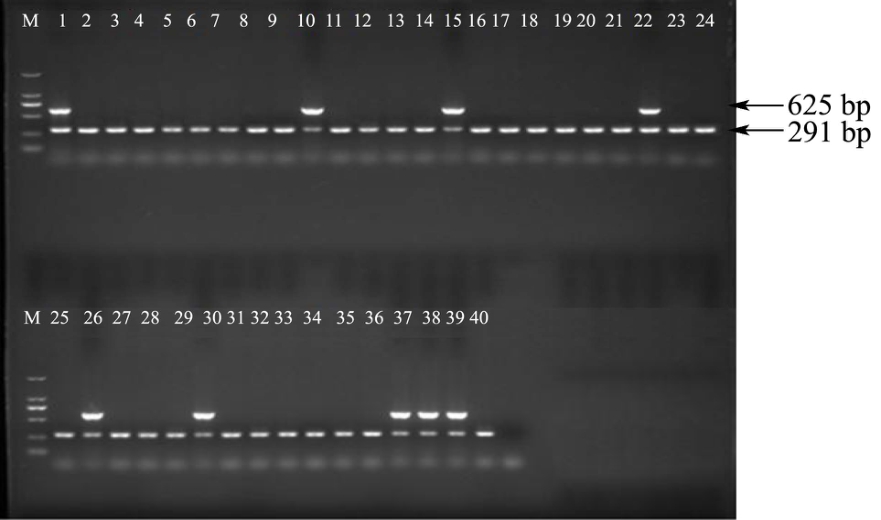
39株大肠杆菌O157:H7菌株经双重PCR检测,有8株鉴定为O157:H7,31株为O157(如图1)。
2.2 毒力基因分布
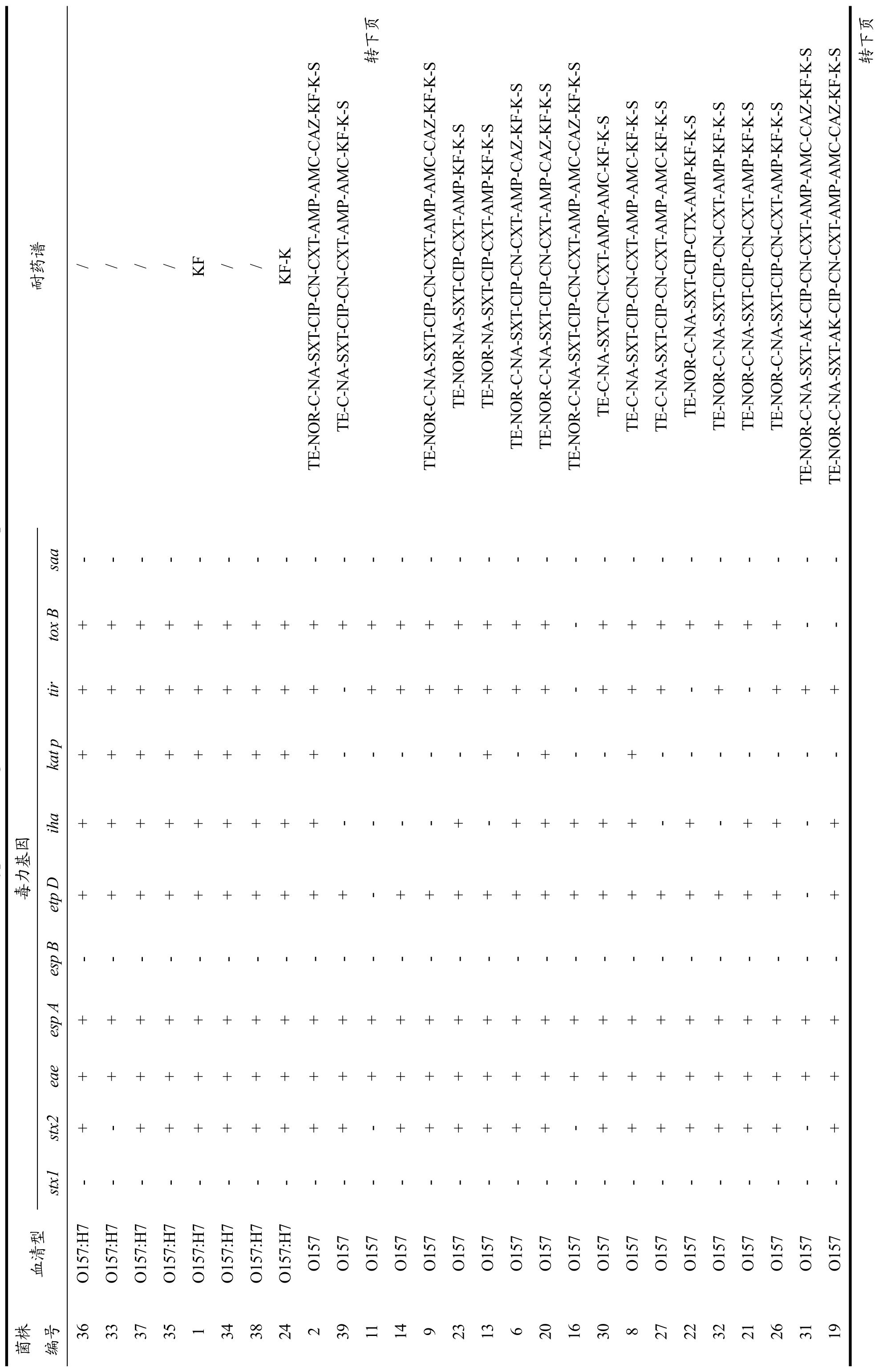
11种毒力基因的分布情况如表2和图2所示。所有菌株都携带eae基因,82.05%携带stx2基因,所有菌株均未携带 stx1基因;大部分菌株含有 espA(92.31%)、etpD(94.87%)、tir(87.18%)、toxB(79.49%)和 iha(69.23%)基因;46.15%菌株含有katP基因。espB和saa基因未检测到。菌株的毒力谱是多样的,大部分菌株携带3种以上的毒力基因,部分菌株携带8种以上毒力基因。
2.3 药敏实验
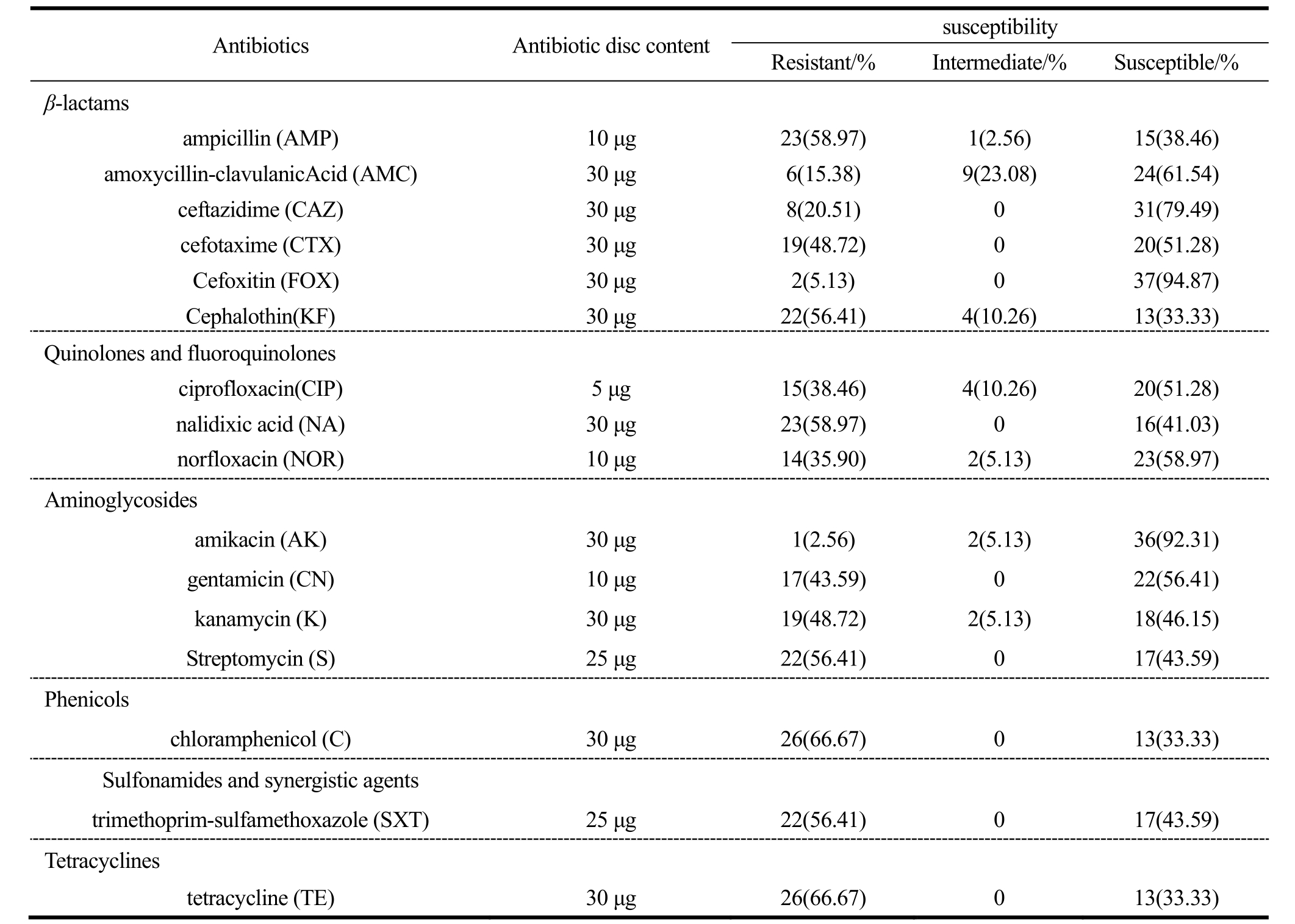
药敏试验结果表明,菌株对 TE、C、SXT、S、NA、KF和AMP具有较高的抗性,对CTX、CN、K、CIP和NOR抗性非常普遍,20.51%菌株对CAZ具有抗性;而对AMC、FOX和AK是相抗性较低(表3)。从抗性谱分布情况来看,30%(30/39)菌株抗三种以上抗生素,为多重耐药菌株;20株(51.28%)菌株抗10~15种抗生素(表2)。
表2 菌株血清型、毒力谱和耐药谱分布
Table 2 The serotypes, virulence genes, and antimicrobial resistance profiles of 39 isolates
?
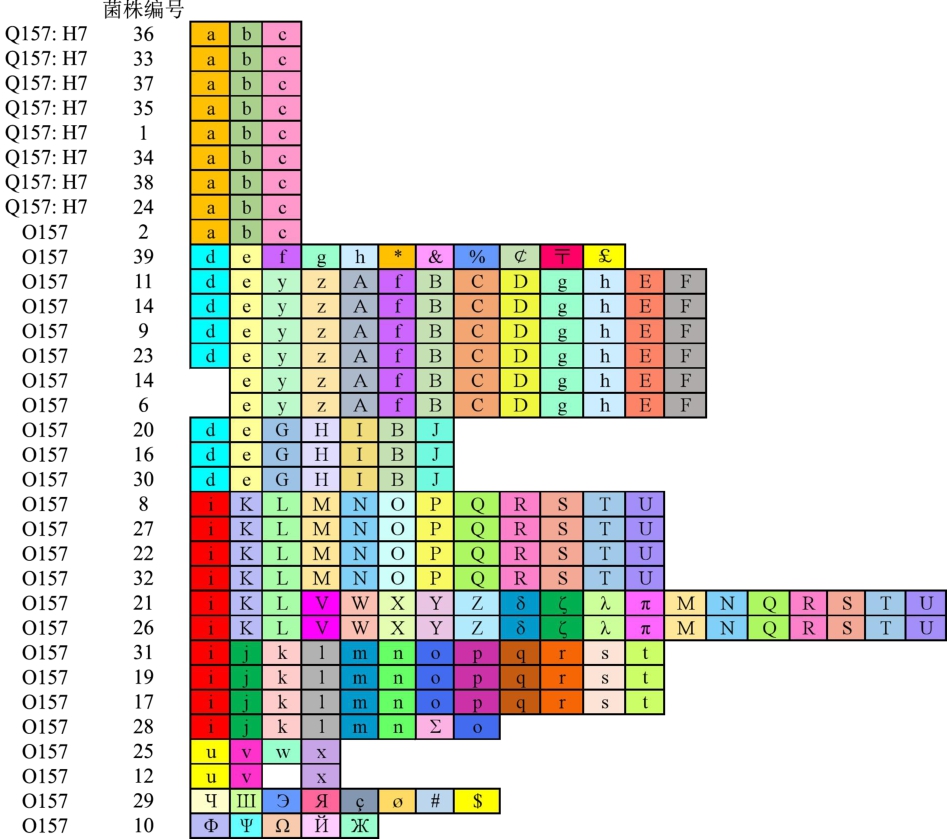
接上页
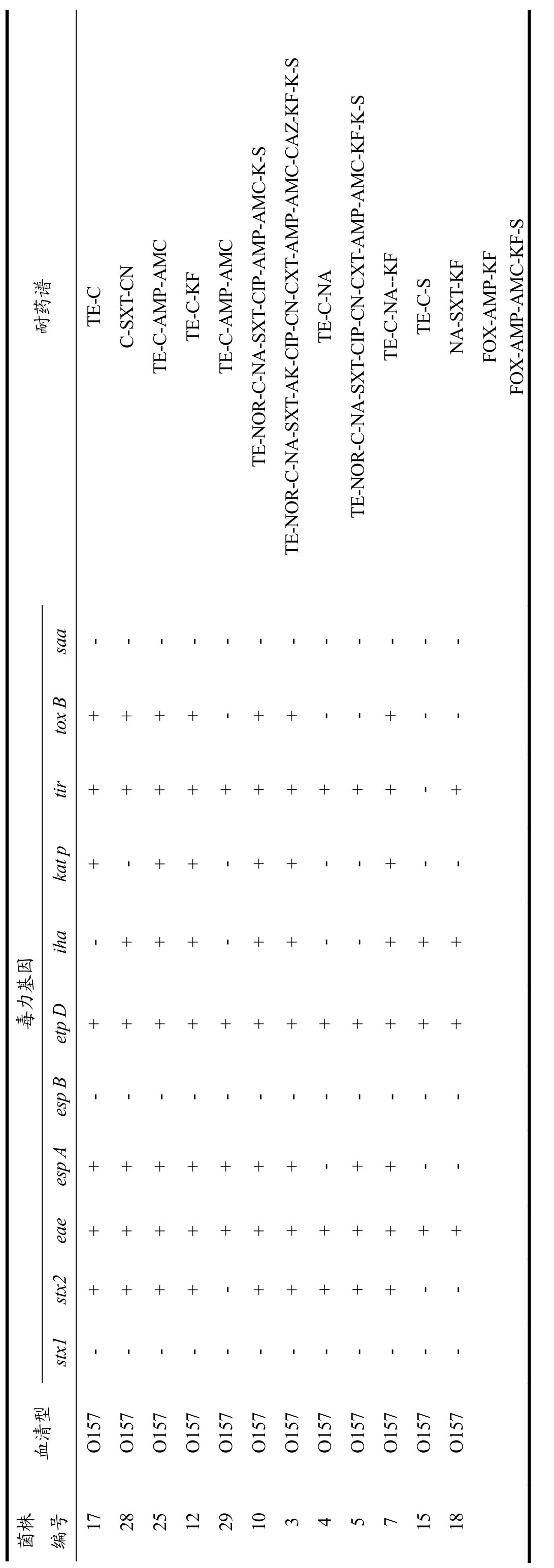
?
表3 菌株的抗生素抗性结果
Table 3 Antimicrobial susceptibility of 39 isolates
Antibiotics Antibiotic disc content susceptibility Resistant/% Intermediate/% Susceptible/%β-lactams ampicillin (AMP) 10 μg 23(58.97) 1(2.56) 15(38.46)amoxycillin-clavulanicAcid (AMC) 30 μg 6(15.38) 9(23.08) 24(61.54)ceftazidime (CAZ) 30 μg 8(20.51) 0 31(79.49)cefotaxime (CTX) 30 μg 19(48.72) 0 20(51.28)Cefoxitin (FOX) 30 μg 2(5.13) 0 37(94.87)Cephalothin(KF) 30 μg 22(56.41) 4(10.26) 13(33.33)Quinolones and fluoroquinolones ciprofloxacin(CIP) 5 μg 15(38.46) 4(10.26) 20(51.28)nalidixic acid (NA) 30 μg 23(58.97) 0 16(41.03)norfloxacin (NOR) 10 μg 14(35.90) 2(5.13) 23(58.97)Aminoglycosides amikacin (AK) 30 μg 1(2.56) 2(5.13) 36(92.31)gentamicin (CN) 10 μg 17(43.59) 0 22(56.41)kanamycin (K) 30 μg 19(48.72) 2(5.13) 18(46.15)Streptomycin (S) 25 μg 22(56.41) 0 17(43.59)Phenicols chloramphenicol (C) 30 μg 26(66.67) 0 13(33.33)Sulfonamides and synergistic agents trimethoprim-sulfamethoxazole (SXT) 25 μg 22(56.41) 0 17(43.59)Tetracyclines tetracycline (TE) 30 μg 26(66.67) 0 13(33.33)
2.4 CRISPR分子分型结果
利用PCR扩增菌株的CRISPR1位点,结果发现39株菌株中有33株(84.61%)含有CRISPR1位点,其中8株O157:H7全部含有CRISPR1位点,31株O157中 26株检测到 CRISPR1位点,6株未检测到,CRISPR1位点的PCR扩增产物符合其长度。
将33株菌株的spacer信息与CRISPR database数据库的CRISPR序列用CRISPR Finder软件进行分析,结果显示菌株中含有两条保守的 29 bp的共有序列DR序列(CGGTTTATCCCCGCTGGCGCGGGGAAC AC和CGGTTTATCCCCGCTGGCGCGGGGAACT C);33株菌株共产生了76条独特的spacer序列(暂未列出),每个菌株spacer数量为3~19个。将spacer序列进行排列后,产生的图谱结果如图3所示,从图中可以看出,菌株具有较高的遗传多态性。8株O157:H7 CRISPR spacer位点分布完全一致。26株O157菌株呈现13种不同的spacer图谱。
3 讨论与结论
3.1 大肠杆菌O157导致的食源性疾病已引起了人们的高度重视,但目前我国食品源O157菌株的病原特点等方面的研究仍相对较少,对其毒力因子分布、耐药变化和遗传特性缺乏系统的调查数据。前期研究中,尽管已有学者对大肠杆菌O157的毒力因子进行了研究,但仅限于少数几个毒力基因如eae、stx和hly,而对其他毒力因子分布情况未见详细报道,且其研究区域多为某个地区或省份。水新云等[18]从武汉市售肉类食品中分离到4株O157:H7菌株,检测发现这4株菌都携带eae、stx和hly基因。白莉等[19]对2005~2007年中国食品中疑似大肠杆菌O157进行四个毒力基因(eae、hlyA、stx1和stx2)检测,发现菌株出现多种不同毒力基因谱。Sallam等[20]对非洲零售牛肉中分离的大肠杆菌O157和O157:H7进行了四种毒力基因检测(eae、hlyA、stx1和stx2),发现46.70%菌株同时含有eae、hlyA、stx1和stx2,40%菌株同时含有eae、stx2和hlyA基因。Dong等[21]对我国牛肉加工厂分离的O157:H7的毒力基因进行鉴定,结果发现eae、stx2和hlyA基因存在于所有菌株中,而stx1基因未检测到。赵嘉咏[22]对河南省2009~2010年收集的O157进行了eae和hly毒力基因检测,发现来自食品的8株O157均不含毒力因子。
3.2 本研究同时对11种毒力基因进行了检测,eae,stx1和stx2携带率与文献报道有所不同,分析可能与样品来源和地区差异有关。另外,不同年份分离的菌株其毒力特点也有不同,表明菌株可能会随时间变化而发生变异。除了eae、stx1和stx2基因,本研究还对LEE毒力岛和大质粒(pO157)上的其他毒力基因进行了研究,发现espA katP、toxB、tir和etpD等广泛存在于O157和O157:H7菌株中,这些毒力因子已被证实与致病性相关。因此,加强其他毒力因子的监测非常必要。
3.3 药敏实验结果发现,本次分离的菌株对临床和农业上常用的抗生素产生了较高的抗性,特别是对TE、SXT、NA、S、C、KF和 AMP,这与文献报道的具有相似之处。Msolo等[23]调查了南非某省原料奶、奶制品机械和工人手套等样品分离的 O157:H7的耐药性,发现菌株对TE(81%)、S(52%)和C(45%)高度耐药。Disassa等[24]对从原料牛奶分离的O157:H7进行耐药研究,发现菌株对TE(81.8%)、S(81.8%)和 K(63.6%)具有高度抗性。Beyi[25]对肉店和餐馆分离的大肠杆菌 O157耐药性进行检测,结果发现 9株中5株抗AMC、2株抗S和3株抗C。分析可能与该国临床和畜牧业中长期大量使用这类抗生素有关。从耐药谱来看,超过30%菌株具有多重耐药性。部分菌株对CAZ和CTX有一定抗性,可能为超广谱抗性菌株。由于抗性菌株可通过食物进入人体,给人类健康带来很大隐患,因此需要密切跟踪这些菌株的变化,减少其传播和扩散。
3.4 本研究采用CRISPR1位点对菌株的遗传特点进行了分析,结果发现菌株具有较高的遗传多样性。即使同一血清的菌株也可能出现不同的遗传进化特征,利用CRISPR分型可以深入分析其变异特点。值得说明的是,本研究中有部分菌株缺失CRISPR1位点,这与之前文献报道有一致之处。Ison等[26]对牛粪中的大肠杆菌 O26:H11菌株进行 CRISPR分型,发现CRISPR1和CRISPR2位点有不同程度缺失。Tymensen等[27]用 CRISPR1分析了地表水和粪便样品中的大肠杆菌,结果有菌株CRISPR1位点未能检测到。在沙门氏菌 CRISPR分子分型方面,庄孝飞[28]利用设计的CRISPR1引物对36种沙门氏菌血清型进行PCR扩增,结果发现5种血清型未能扩增出目的条带,无法分型。这些结果表明,尽管CRISPR分型具有高的分辨率,但是单一的分子分型技术仍不够完善,需要辅助其他分型技术,多种分子分型技术综合应用会更有利深入挖掘生物学特点。另外,本研究未发现毒力谱和耐药性与CRISPR分型结果间的明显相关性,可能与菌株数量少有关,后续工作中可机进一步深入研究。
参考文献
[1] Galli L, Brusa V, Singh P, et al. High prevalence of clade 8 Escherichia coli O157:H7 isolated from retail meat and butcher shop environment [J]. Infection Genetics and Evolution, 2016, 45: 1-5
[2] D'Astek B A, DEL-Castillo L L, Miliwebsky E, et al.Subtyping of Escherichia coli O157:H7 strains isolated from human infections and healthy cattle in Argentina [J].Foodborne Pathogens and Disease, 2012, 9(5): 457-464
[3] Sharapov U M, Wendel A M, Davis J P, et al. Multistate outbreak of Escherichia coli O157:H7 infections associated with consumption of fresh spinach: United States, 2006 [J].Journal of Food Protection, 2016, 79(12): 2024-2030
[4] 陈雅君,王亚宾,张莉娟,等.动物源性肠出血性大肠杆菌O157:H7及其3个毒力基因的多重PCR快速检测研究[J].中国人兽共患病学报,2013,29(7):686-691 CHEN Ya-jun, WANG Ya-bin, ZHANG Li-juan, et al.Detection of animal-derived Escherichia coli O157:H7 and its three virulence genes by multiplex PCR technique [J].Chinese Journal of Zoonoses, 2013, 29(7): 686-691
[5] 张书萧.大肠杆菌 O157的分子流行病学调查和毒力因子研究[D].长春:吉林农业大学,2012 ZHANG Shu-xiao. Molecular epidemiology and study on virulence factor of Escherichia coli O157 [D]. Changchun:Jilin Agricultural University, 2012
[6] 万成松.大肠杆菌O157:H7的毒力岛与毒力因子的研究进展[J].微生物学免疫学进展,2006,34(2):58-62 WAN Cheng-song. The pathogenic islands and virulence factors of Escherichia coli O157:H7 [J]. Progress in Microbiology and Immunology, 2006, 34(2): 58-62
[7] Kim H S, Chon J W, Kim Y J, et al. Prevalence and characterization of extended-spectrum-β-lactamaseproducing Escherichia coli, and Klebsiella pneumoniae, in ready-to-eat vegetables [J]. International Journal of Food Microbiology, 2015, 207: 83-86
[8] Yang B, Wang Q, Cui S, et al. Characterization of extended-spectrum beta-lactamases-producing Salmonella strains isolated from retail foods in Shaanxi and Henan province, China [J]. Food Microbiology, 2014, 42(12): 14-18[9] Ahmed A M, Shimamoto T. Molecular analysis of multidrug resistance in Shiga toxin-producing Escherichia coli O157:H7 isolated from meat and dairy products [J].International Journal of Food Microbiology, 2015, 193: 68-73
[10] 叶菊莲,占利,梅玲玲,等.利用 MLST技术对浙江省大肠杆菌 O157的分子流行病学研究[J].中国人兽共患病学报,2011,27(10):901-904 YE Ju-lian, ZHAN Li, MEI Ling-ling, et al. Multilocus sequence typing analysis application in molecular epidemiologicalresearch of Escherichia coli O157 isolates in Zhejiang province [J]. Chinese Journal of Zoonoses, 2011,27(10): 901-904
[11] 王开,裴志花,陈中秋,等.常见食源性致病菌及其分子分型技术的研究进展[J].中国兽医杂志,2013,49(12):51-54 WANG Kai, PEI Zhi-hua, CHEN Zhong-qiu, et al. Research progress of common food-borne pathogens and their molecular typing techniques [J]. Chinese Journal of Veterinary Medicine, 2013, 49(12): 51-54
[12] Fabre L, Zhang J, Guigon G, et al. CRISPR typing and subtyping for improved laboratory surveillance of Salmonella infections [J]. PLoS One. 2012, 7(5): e36995
[13] Shariat N, Dudley E G. CRISPRs: molecular signatures used for pathogen subtyping [J]. Applied and Environmental Microbiology, 2014, 80(2): 430-439
[14] Di H, Ye L, He Y, et al. Comparative analysis of CRISPR loci in different Listeria monocytogenes, lineages [J].Biochemical and Biophysical Research Communications,2014, 454(3): 399-403
[15] 梁文娟,张荣光,段广才,等.基于 CRISPR 对大肠埃希菌O157:H7的检测[J].西安交通大学学报(医学版),2016,37(5):748-753 LIANG Wen-juan, ZHANG Rong-guang, DUAN Guang-cai,et al. Detection of Escherichia coli O157:H7 based on CRISPR locu [J]. Department of Preventive Medicine, Henan University of Science and Technology, 2016, 37(5): 748-753
[16] Monaghan Á, Byrne B, Fanning S, et al. Serotypes and virulence profiles of atypical enteropathogenic Escherichia coli, (EPEC) isolated from bovine farms and abattoirs [J].Journal of Applied Microbiology, 2013, 114(2): 595-603
[17] Paton A W, Paton J C. Detection and characterization of Shiga toxigenic Escherichia coli by using multiplex PCR assays for stx1, stx2, eaeA, enterohemorrhagic E. coli hlyA,rfbO111, and rfbO157 [J]. Journal of Clinical Microbiology,1998, 36(2): 598-602
[18] 水新云,王虎虎,高峰,等.4株E. coli O157:H7毒力基因检测及其冷应激损伤[J].食品科学,2016,37(4):176-180 SHUI Xin-yun, WANG Hu-hu, GAO Feng, et al. Detection of virulence genes of four Escherichia coli O157:H7 strains and cold stress injury in the bacteria [J]. Food Science, 2016,37(4): 176-180
[19] 白莉,刘秀梅,付萍,等.2005~2007年中国食品中疑似 O157大肠埃希菌的鉴定及毒素基因的检测[J].卫生研究,2010,39(3):335-338 BAI Li, LIU Xiu-mei, FU Ping, et al. Serotyping and virulence genes of suspected Escherichia coli O157 strains in food from 2005 to 2007 [J]. Journal of Hygiene Research,2010, 39(3): 335-338
[20] Sallam K I, Mohammed M A, Ahdy A M, et al. Prevalence,genetic characterization and virulence genes of sorbitol-fermenting Escherichia coli O157:H- and E. coli O157:H7 isolated from retail beef [J]. International Journal of Food Microbiology, 2013, 165(3): 295-301
[21] Dong P, Zhu L, Mao Y, et al. Prevalence and characterization of Escherichia coli O157:H7 from samples along the production line in Chinese beef-processing plants [J]. Food Control, 2015, 54(1): 39-46
[22] 赵嘉咏,穆玉姣,张白帆,等.河南省2009-2010年出血性大肠埃希菌O157毒力基因分布与脉冲场凝胶电泳分型研究[J].中华流行病学杂志,2015,36(11):1324-1326 ZHAO Jia-yong, MU Yu-jiao, ZHANG Bai-fan, et al.Distribution of virulence genes and PFGE molecular typing of Entero-hemorrhagic Escherichia coli O157 in Henan from 2009 to 2010 [J]. Chinese Journal of Epidemiology, 2015,36(11): 1324-1326[20] 王薇薇.红松松塔多糖的提取、分离纯化及抗氧化活性研究[D].青岛:青岛科技大学,2013 WANG Wei-wei. Study on the extracion,purification and anti-oxidation activities of polysaccharide from pine cone of Pinus koraiensis [D]. Qingdao: Qingdao University of Science & Technology, 2013
[21] Guo F F, Chen D Y, Dong L, et al. Study on extraction and antioxidant activity of polysaccharide from the pine cone of Pinus tabulieformis carr [J]. Ginseng Research, 2014
[22] 张根生,侯静,张铭东,等.松仁红衣多酚的提取及体外抗氧化活性研究[J].食品工业科技,2014,35(23):252-256 ZHANG Gen-sheng, HOU Jing, ZHANG Ming-dong, et al.Research of extraction and antioxidant activity in vitro of polyphenols from pine nut coat [J]. Science and Technology of Food Industry, 2014, 35(23): 252-256
[23] Yingbin Shen, Hui Zhang, Lilin Cheng, et al. In vitro and in vivo antioxidant activity of polyphenols extracted from black highland barley [J]. Food Chemistry, 2016, 194: 1003-1012
[24] 马承慧,王群,刘牧.3种松科植物松针多酚的体外抗氧化活性评价[J].西南农业学报,2016,29(5):1063-1068 MA Cheng-hui, WANG Qun, LIU Mu. Evaluation of antioxidant activity of pine polyphenols from three pinaceae species in vitro [J]. Southwest China Journal of Agricultural Sciences, 2016, 29(5): 1063-1068
[25] 黄琴,沈杨霞,张成静,等.铁皮石斛多酚和黄酮含量及与抗氧化活性的相关性[J].应用与环境生物学报,2014, 20(3):438-442 HUANG Qin, SHEN Yang-xia, ZHANG Cheng-jing, et al.Correlation of the antioxidant property with the total phenolic content and total flavonoids of different Dendrobium officinale extracts [J]. Chinese Journal of Applied and Environmental Biology, 2014, 20(3): 438-442
[26] Du G, Li M, Ma F, et al. Antioxidant capacity and the relationship with polyphenol and vitamin C in Actinidia fruits[J]. Food Chemistry, 2009, 113(2): 557-562
[27] Davide T, Elena V, Davide B, et al. In vitro bio-accessibility and antioxidant activity of grape polyphenols [J]. Food Chemistry, 2010, 120(2): 599-606
The Virulence Genes, Antimicrobial Susceptibility, and CRISPR Molecular Typing of Escherichia coli O157 from Retail Foods in China
YANG Guang-zhu, ZHANG Shu-hong, HUANG Yuan-bin, WU Qing-ping, ZHANG Ju-mei
(State Key Laboratory of Applied Microbiology Southern China, Guangdong Provincial Key Laboratory of Microbial Culture Collection and Application, Guangdong Open Laboratory of Applied Microbiology, Guangdong Institute of Microbiology, Guangzhou 510070, China)
Abstract: Escherichia coli O157 is an important food-borne pathogen. 39 E. coli O157 strains identified during 2014 and 2015 in retail foods in china were identified by double PCR to understand the biological characteristics of food-borne E. coli O157. The virulence genes and antimicrobial susceptibility of isolates were also detected, and the genetic diversity of Escherichia coli O157 was analyzed by Clustered Regularly Interspaced Short Palindromic Repeat (CRISPR) typing. The results showed that 8 were identified as O157:H7 and 31 were O157 among the 39 strains. , Eae gene was detected in all the isolates, and stx2 was in 82.50% strains of isolates, while stx1 was not detected in the tested 11 virulence genes. Other virulence genes such as espA, etpD, tir, toxB, iha, and katP were detected with the carrying rate of 92.31%,94.87%, 87.18%, 79.49%, 69.23%, and 46.15%, respectively. Antimicrobial susceptibility results indicated that the isolates strains were highly resistant to TE, SXT, S, C, and AMP. More than 30% of the isolates were resistance to three and more antibiotics. The CRISPR typing indicated that these isolates strains had a high genetic diversity. Thirty-three of the 39 strains had CRISPR1 loci, including 8 E. coli O157:H7 and 26 E.coli O157. The 8 E. coli O157:H7 produced the same spacer maps, while 26 E. coli O157 produced 13 diversified spacer spectra. This study can provide important basic data for food borne disease surveillance, disease traceability and epidemiological research.
Key words: Escherichia coli O157; virulence genes; antimicrobial susceptibility; Clustered Regularly Interspaced Short Palindromic Repeat (CRISPR) typing
收稿日期:2017-07-04
基金项目:“十三五”国家重点研发计划项目(2016YFD0401204);广东省科技计划项目(2016A040403088、201704020089)
作者简介:杨广珠(1991-),女,研究实习员,研究方向:食源性致病菌检测技术
通讯作者:吴清平(1962-),男,研究员,研究方向:食品微生物安全检测及控制技术
文章篇号:1673-9078(2017)12-29-37
DOI: 10.13982/j.mfst.1673-9078.2017.12.005