
图1 pPICZb-EPSPS重组载体构建流程图
Fig.1 The construction of pPICZb-EPSPS vector
摘要:为建立一种新的外源蛋白安全评估方法,构建转外源基因酵母是关键步骤。本研究以来源于转基因作物的抗除草剂合成酶蛋白CP4-EPSPS为研究模板,首先通过基因优化和化学合成获得cp4-epsps基因,优化后基因密码子适用性指数(CAI)为0.85,GC含量为52%。目的基因克隆至毕赤酵母表达载体pPICZb,经鉴定筛选正确的重组载体pPICZb-EPSPS电击转化至毕赤酵母GS115,cp4-epsps基因通过同源重组方式整合至酵母基因组,PCR扩增酵母基因组筛选得到在正确位点发生同源重组的4株阳性菌株。随后,各阳性菌株分别于28 ℃、250~300 r/min培养,0.5%甲醇诱导4 d后,提取各组菌株总蛋白,采用SDS-PAGE和western blotting鉴定CP4-EPSPS表达的正确性。CP4-EPSPS单克隆抗体及载体标签C-MYC单克隆抗体的western blotting结果一致显示cp4-epsps基因在毕赤酵母GS115中成功获得表达,大小约50 ku。本实验成功构建了转cp4-epsps基因的毕赤酵母基因工程菌,为下一步开展转基因作物CP4-EPSPS成份的安全评价奠定基础。
关键词:转基因作物;外源蛋白;安全评价;cp4-epsps基因;毕赤酵母
转基因(transgenic modified,GM)作物是农业领域的一项重要科技成果,对于粮食实现增产有重要作用[1]。中国在棉花、水稻和玉米等作物的转基因研究方面已形成了自己的特色和优势[2]。然而,由于目前转基因成份诱发的非预期效应不明确,安全评价结果争议大。目前全球商业化种植的转基因作物常见性状之一是抗除草剂,抗除草剂基因中以来源于土壤农杆菌(Agrobacterium sp.)CP4 株系的 epsps(5-enolpyruvyl shikimate-3-phosphate synthesis,cp4-epsps)合成酶基因的使用最为广泛,该酶对除草剂草甘膦有高抗性。随着基因工程技术的发展,也衍生出多种修饰的cp4-epsps转基因作物[3]。由于该基因主要用于大豆玉米等食用性作物,伴随产生的食品安全问题也引起人们极大的关注。
目前该转基因作物食用性安全评价方法是由欧洲食品安全机构(European Food Safety Authority,EFSA)提出,必须对啮齿类动物进行为期90 d的喂食观察,如果90 d内动物连续食用该转基因食物,其各项生理生化指标未显异常,则被认为安全[4]。很多研究按照上述方法评价了多种转基因作物的安全性,结果显示“90 d内无明显数据表明该转基因作物对鼠(猪)有害”[5~12]。与此同时,另一些科学家在此基础上延长喂食时间或增加转基因成份CP4-EPSPS的浓度后发现,小鼠的淋巴细胞、肠抗感染能力和肝细胞代谢率等多项检测指标出现波动[13~16],并且有研究观察期长达 2年,但结果体内代谢差异不明显,更无法确定这些变化是否与毒性相关[17]。另外据调查发现[18,19],国内消费者对转基因作物的最大担忧就是非预期效应。由此可见,改良转基因成份如CP4-EPSPS等的安全评价工作,减少争议结果是转基因作物安全性评价工作中最重要的一环。
但就目前的评价方法和评价结果来看,还存在几点不足:(1)目前研究方式使得动物暴露于外源蛋白的有效浓度太低,动态代谢浓度差异不明显,非预期效应不易被激发,无法深入研究;(2)实验耗时长达两年,在长期喂养中因各种原因导致大鼠死亡,最后无法结合病理检测分析代谢数据,因而仍然无法判断是否存在非预期效应;(3)转基因作物缺乏严格的非转基因作物作为阴性对照。
针对上述问题,新的评价方法要解决以下几个问题:(1)能放大转基因蛋白的暴露浓度,继而能放大动态代谢变化;(2)缩短评价周期,建立筛选平台,能同时对大量转基因作物的外源基因或蛋白进行评价。
通过毕赤酵母表达获的重组蛋白与高等动植物细胞表达系统获得蛋白具有相似的结构和生物活性。本实验选择pPICZb诱导型载体来实现重组蛋白在酵母胞内的可控表达。构建的转cp4-epsps基因酵母工程菌具有以下特点:(1)安全性高。大量研究表明毕赤酵母表达系统被广泛用于人源性疫苗研究[20,21],喂食动物不会产生毒性反应;(2)剂量可控。可以控制酵母细胞的喂食浓度(OD值),提高单位时间内转基因成份CP4-EPSPS的喂食浓度,使得动物暴露于外源蛋白的浓度得以放大,有利于体内代谢浓度差异放大;(3)有严格的非重组酵母菌株作为转 cp4-epsps基因酵母的阴性对照;(4)与构建转基因作物相比,构建转基因酵母简单易操作[22,23],能在短时间内同时构建多种外源基因蛋白重组酵母菌株,便于进一步的评价工作。因此期望构建高通量和短周期的外源蛋白评价平台,成功构建转cp4-epsps基因酵母是关键第一步。
毕赤酵母(Pichia pastoris)GS115菌株及表达载体pPICZb均购自Invitrogen;大肠杆菌DH5α菌株由本实验室保存;cp4-epsps基因由苏州金唯智生物科技有限公司优化合成;EcoRV、HindIII和SacI限制性内切酶、PrimeSTAR HS高保真聚合酶(R010Q)、In-Fusion HD (639633)等购至大连宝生物工程公司;质粒小量提取试剂盒购自 Sigma-Aldrich公司;抗CP4-EPSPS单克隆抗体购自 Fitzgerald(货号10R-10541);CP4-EPSPS转基因作物的检测试纸条由农业部转基因植物环境安全监督检测测试中心惠赠;C-MYC单克隆抗体和Rabbit anti-mouse IgG-HRP均购自Santa Cruz;PVDF膜购自Merck Millipore。
1.2.1 pPICZb-EPSPS重组表达载体的构建
根据Genbank KP212901.1序列设计cp4-epsps基因,并根据毕赤酵母 Pichia pastoris偏好性优化cp4-epsps基因密码子,同时使 GC含量保持于30~70%,基因设计同时考虑 Kozak序列(GCCACCAUGG)和同源重组位点 sfuI (TTCGAA)和XbaI(TCTAGA)。优化软件OPITIMIZE由苏州金唯智生物科技有限公司提供,同时化学合成获得cp4-epsps基因,PrimeSTAR HS高保真聚合酶扩增目的基因,反应体系为:pUC57-EPSPS质粒(250 ng/mL)1 μL,5×Buffer(Mg2+)10 μL,dNTP Mixture(各 2.5 mM)4 μL,上下游引物(20 pmol/μL)各0.5 μL,DNA聚合酶0.5 μL。反应条件为 98 ℃ 10 s,55 ℃ 10 s,72 ℃ 1 min,30循环。pPICZb和目的基因分别采用用sfuI和XbaI酶切纯化,采用In-Fusion HD克隆试剂盒将cp4-epsps基因整合至pPICZb载体,构建pPICZb-EPSPS重组载体,反应体系为pPICZb(50 ng/μL)1 μL,目的基因(约 80 ng/μL)1 μL,5×In-Fusion HD Enzyme Premix 2 μL,加dH2O至10 μL,50 ℃反应10 min,随后取1 μL转化E.coli DH5α感受态,筛选提取阳性克隆质粒,进一步鉴定载体的正确性。
1.2.2 pPICZb-EPSPS重组载体的鉴定
提取pPICZb-EPSPS重组质粒,使质粒终浓度为250 ng/μL。采用EcoRⅤ和HindIII双酶切鉴定该重组质粒,反应体系为 20 μL (ddH2O 12 μL,10×Quickcut buffer 2 μL,EcoRV 0.5 μL,HindIII 0.5 μL,pPICZb-EPSPS载体5 μL,37 ℃反应20 min,电泳鉴定正确的菌株,提取质粒用于测序(苏州金唯智生物科技有限公司)鉴定cp4-epsps基因序列有无突变,是否与理论设计序列完全一致。
1.2.3 转 cp4-epsps重组毕赤酵母的筛选及鉴定
根据 Invitrogen公司提供载体 pPICZb克隆手册(货号:V190-20)完成重组毕赤酵母的筛选。
电击转化:采用YPD培养基培养菌株GS115至OD600约1.3~1.5时收集细胞,采用预冷1 mol/L山梨糖醇重新溶解悬浮该酵母细胞,以制备酵母感受态备用;SacI酶切线性化已重组载体 pPICZb-EPSPS,电击转化至酵母感受态细胞中,电压1800 V,瞬时电击后取出,立即加入1 mL预冷山梨糖醇并置于30 ℃孵化 2~4 h后取 200 μL涂布于含有 Zecoin浓度(50 μg/mL)的YPDS平板,30 ℃中培养2~3 d,直至菌落形成。挑选10~20个菌落至新鲜YPD(Zecoin+)液体培养基中,30 ℃培养过夜,提取酵母总基因组采用PCR鉴定目的基因的存在,得到的阳性菌株分别编号。
PCR 检测:25 μL体系(ddH2O 17 μL,dNTP Mix 2 μL,10×PCR buffer 2.5 μL,5’AOX1引物1 μL,3’AOX1引物 1 μL,总基因组 1 μL,Taq 聚合酶 0.5 μL),94 ℃1 min,55 ℃ 1 min,72 ℃ 1.5 min,25循环。各取5 μL反应液进行凝胶电泳。
1.2.4 cp4-epsps基因在酵母中的表达
分组:空白对照组A为宿主细胞菌株GS115,阴性对照组B为转化空载体pPICZb重组酵母菌株以及实验组C为转化重组载体pPICZb-EPSPS的重组酵母菌株。
根据 Invitrogen公司提供毕赤酵母表达手册(货号:K1740-01)完成重组毕赤酵母诱导表达。
诱导表达:分别将空白对照组A、阴性对照组B和实验组C酵母菌株接种至含有25 mL BMGY培养基中,28~30 ℃、250~300 r/min 恒温振荡培养16~18 h至OD600约4,收集细胞,弃上清,用新鲜的BMMY培养基100~200 mL重悬菌体沉淀到OD600为1.0,然后各组分别取50 mL重悬液转移至250 mL三角瓶中,28~30 ℃、250~300 r/min开始诱导表达:每隔24 h补加100%甲醇至终浓度为0.5%,诱导培养4 d后,各组分别取菌液12000 r/min离心,检测OD600,离心5 min,待提取蛋白。
蛋白提取:每 20 OD600样品离心去上清后加入150 μL裂解缓冲液重悬,加等量酸洗玻璃珠(0.5 mm),涡旋30 s,冰上孵育30 s,重复8次,4 ℃离心10 min,将上清转移至干净的微量离心管中,取50 μL上清,加等体积2×SDS上样缓冲液煮沸,待蛋白电泳。
1.2.5 CP4-EPSPS蛋白的western blotting鉴定
采用SDS-PAGE分离蛋白样品,同时PVDF膜采用甲醇预处理3~5 s,放至转印液浸润半小时。电泳结束后取出凝胶,通过电转移将蛋白转移至PVDF膜,低温操作,100 V恒压60~120 min,取出杂交膜,TBST漂洗5 min三次,5%脱脂奶粉溶液室温封闭1 h,TBST洗膜三次,加入CP4-EPSPS单克隆抗体(1:500) 4 ℃过夜,TBST洗膜三次,Rabbit anti-mouse IgG-HRP(1:5000)稀释液37 ℃孵育1 h,TBST洗膜,放至暗盒,显影。

图1 pPICZb-EPSPS重组载体构建流程图
Fig.1 The construction of pPICZb-EPSPS vector
图1 为pPICZb-EPSPS重组载体构建流程图。其中化学合成的cp4-epsps基因首先克隆在pUC57载体上。pPICZb载体大小为3328 bp,载体上SfuI和XbaI位点为 cp4-epsps基因的同源重组位点,HindIII和EcoRV位点为pPICZb-EPSPS重组载体的鉴定位点。
为提高mRNA的稳定性及蛋白表达效率[24,25],须根据毕赤酵母细胞 Pichia pastoris的偏好性进行cp4-epsps基因密码子优化,减小稀有密码子的使用,提高最优密码子在基因序列中的比例,同时使基因的GC含量保持在30~70%之间,丰富的GC含量可大大提高该基因的表达水平[26],低水平GC含量将限制基因转录[26,27]。基因优化结果见图2和图3,优化序列见附件1。

图2 密码子适用性指数
Fig.2 Codon Adaptation Indexes (CAI)
注:该图为基因序列各密码子的利用率分布图。一般认为当CAI为1.0时,基因表达水平最为理想,CAI值越高,其表达水平越高。

图3 不同利用率密码子在基因中的百分含量
Fig.3 Frequency of Optimal Codons (FOP)
图2 为基因序列中各密码子的利用率分布图,由密码子适用性指数(Codon adaption index,CAI)表征。通常认为,当CAI为1.0时,是菌株表达最理想状态,其表达水平最高,当CAI>0.9时,表达水平理想。图2结果所示,序列经优化后,其CAI值由0.77增加至0.85,表明理论表达水平将得到提高。
图3为不同利用率密码子的百分含量。由图可知,原始序列中利用率在 90~100%的密码子所占比例约50%,序列经优化后,所占比例大于55%,提高了利用率在 90~100%的密码子的含量,同时利用率在70~80%,60~70%和 50~60%的密码子的含量相应提高。序列经优化后,大大降低了稀有密码子的比例。研究表明[25]当最优密码子含量低于40%时,mRNA的平均半衰期为 5.4 min,当最优密码子含量高于70%时,mRNA的平均半衰期为17.8 min,可见密码子的优化对稳定mRNA有重要作用。
经过优化,序列中平均GC含量由最初49%提高至52%。KudlaG等[26]研究比较了同一基因GC丰富序列和GC贫乏序列在哺乳动物细胞中的表达情况,发现GC含量丰富的序列更易进行有效的转录或mRNA的加工处理,GC含量丰富的序列通过提高mRNA的稳定性从而提高基因的表达水平。在基因优化过程中同时还去除A/T或G/C重复序列,避免mRNA形成发夹结构或其它特殊结构,防止mRNA过早终止翻译[28,29]。
最后,基于Genbank KP212901.1 cp4-epsps基因序列(1368 bp),GS115菌株偏好性和载体要求,通过PCR扩增在ATG上游增加Kozak序列和同源重组位点序列和酶切位点,目的基因大小增加至1380 bp。根据Kozak规则[29,30],在基因AUG上游增加Kozak可显著提高转录和翻译效率。
目的基因合成后克隆至pUC57载体上,通过PCR反应获得含有 Kozak序列,同源重组点和 SfuI以及XbaI酶切位点的基因。PCR扩增结果见图3,片段大小与理论结果相符。

图4 cp4-epsps基因扩增
Fig.4 The PCR results of cp4-epsps gene
注:1为cp4-epsps基因扩增条带,M为DL5000。
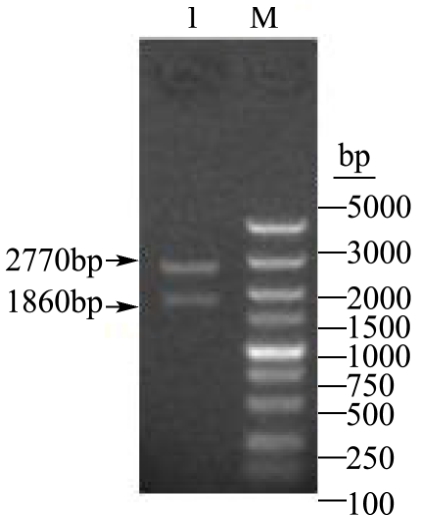
图5 pPICZb-EPSPS载体双酶切鉴定结果
Fig.5 The identification results of pPICZb-EPSPS vector
注:1为HindIII和EcoRV酶切后鉴定结果,M为DL5000。
由图1可知,pPICZb空载体大小为3328 bp,其中HindIII位点位于第873 bp,EcoRV位点位于第2337 bp,当采用HindIII和EcoRV酶切空载体时,产生的两条带大小分别约为1860 bp和1460 bp;当大小为1380 bp的目的片段cp4-epsps整合至位于HindIII和EcoRV位点间的sfuI和xbaI酶切位点后,采用HindIII和EcoRV酶切片段,则产生的两条带大小分别为1860 bp和2770 bp。根据酶切结果(图4),显示cp4-epsps成功克隆至pPICZb载体中,但需进一步测序判断重组载体中目的基因序列与理论设计序列是否一致。测序结果显示与理论相符,无基因突变,结果显示pPICZb-EPSPS载体构建成功。

图6 酵母基因组的PCR鉴定筛选
Fig.6 Selection and identification of genomic DNA derived from yeast strains
注:M为DL5000 marker;泳道5、6、9、10为PCR鉴定的阳性克隆结果,一条带约为2.2 kb,另一条带约为1620 bp,所对应的菌株编号为7,8,11,12号;泳道1、4、7、8、11、12为阴性鉴定结果;泳道2、3为阳性对照,产生一条约2.2 kb的条带。
用SacI单酶切线性化重组质粒pPICZb-EPSPS,载体的线性化可提高基因重组至酵母基因组的机率,而外源基因重组至基因组可提高基因遗传的稳定性。外源基因是否成功重组至基因组,需要通过提取酵母基因组和PCR实验加以验证。cp4-epsps基因导入酵母后,同源重组位点仅发生在AOX1处,在正确位置发生同源重组的转基因酵母可用5’AOX1和3’AOX1通用引物扩增酵母基因组,由于GS115是Mut+型,阳性克隆子的酵母基因组的PCR结果会产生两条带,一条是约2.2 kb的AOX1基因,另一条带略大于目的基因约1620 bp;PCR扩增结果未出现两条带表明为阴性克隆,或重组位点不发生在AOX1处。由图6可判断泳道5、6、9和10对应的酵母菌株为阳性克隆,即目的基因成功整合至酵母基因组,克隆子编号分别为7、8、11和12号。
2.5.1 SDS-PAGE电泳分离重组酵母蛋白

图7 cp4-epsps在酵母中的表达和SDS-PAGE电泳
Fig.7 SDS-PAGE electrophoresis and the expression of the cp4-epsps gene in Pichia pastoris
注:1为阴性酵母GS115蛋白提取物;4为pPICZb空载体转化酵母的蛋白提取物;2、3、5和6分别对应克隆子7、8、11和12的酵母细胞提取物;M为蛋白低分了量marker。
通过SDS-PAGE和western blotting确认cp4-epsps基因在酵母中的表达。成熟的CP4-EPSPS蛋白大小约47.6 ku,pPICZb中C末端的C-MYC标签大小约2.5 ku,通过该载体表达获得的重组蛋白应为CP4-EPSPSMYC融合蛋白,大小应约为 50 ku。由图 7的SDS-PAGE可知,在大约50 ku处有一条与阴性对照和空载体转化酵母对照明显不同的条带,但其特异性还需通过western blotting鉴定。
2.5.2 CP4-ESPSP蛋白的western blotting鉴定
采用CP4-EPSPS蛋白的单克隆抗体进行Western blotting,图8a结果显示在50 ku和小于50 ku处均有特异性条带出现两者进一步通过载体标签C-MYC单克隆抗体的 western blotting结果确定特异性。图 8b结果显示在50 ku附近有特异性条带,与图8a箭头所指条带一致,从而可以判断50 ku的条带为特异性条带,这一结果与理论值相符,从而可以认为目的基因cp4-epsps在毕赤酵母中得到了正确表达。
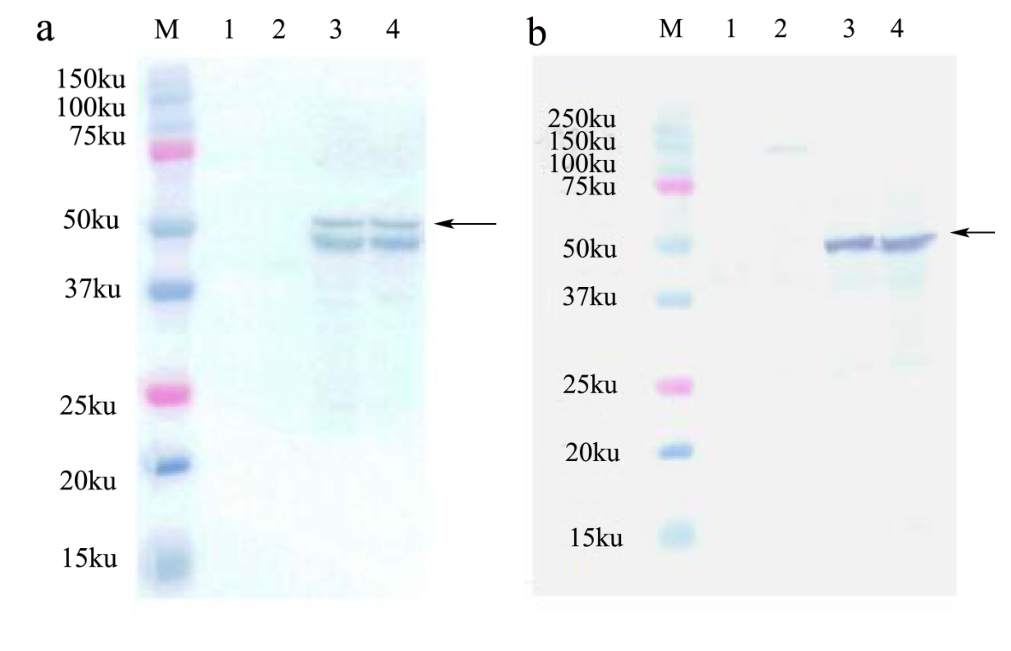
图8 CP4-EPSPS重组蛋白的Western blotting检测
Fig.8 The western blotting results of the CP4-EPSPS recombinant protein
注:a采用 CP4-EPSPS蛋白单克隆抗体进行的 western blotting鉴定结果,有50 ku大小的特异性条带;b采用C-MYC标签单克隆抗体进行的western blotting鉴定结果,有50 ku大小的特异性条带。
图8a中还存在另一明显条带,大小推测约为47 ku,与不带C-MYC标签的蛋白大小相似,也与该基因在原核大肠杆菌表达系统中的表达大小一致[31,32],并且该条件与阴性对照及图8b中结果对比提示,该条带也应为阳性菌株中的特异性条带,推测重组酵母中可能有丢失C-MYC标签的重组 CP4-EPSPS蛋白存在,或有被轻微降解的CP4-EPSPS蛋白存在。
为提高重组蛋白CP4-EPSPS在酵母中的表达量,本实验首先根据毕赤酵母的偏好性,基因序列中最优GC含量、稳定 mRNA结构等要素等优化合成cp4-epsps基因,然后通过同源重组使外源基因整合至酵母基因组,提高基因遗传的稳定性,最后通过CP4-EPSPS单克隆抗体和C-MYC标签单克隆抗体确定CP4-EPSPS表达的特异性条带,从而确认外源基因表达的正确性。至此,可以认为cp4-epsps基因在毕赤酵母中获得了成功表达。本实验室构建的转cp4-epsps基因酵母在后期可以通过浓度控制和外源蛋白的活性控制进一步研究 CP4-EPSPS蛋白剂量与外源蛋白安全性,蛋白活性与外源蛋白安全性的关系,这是后期建立高通量的外源蛋白安全评价平台的关键步骤。
致谢
感谢中国留学基金管理委员会资助(No.201608440425)。
参考文献
[1] James, Clive. 20th Anniversary (1996 to 2015) of the Global Commercialization of Biotech Crops and Biotech Crop Highlights in 2015. ISAAA Brief, 2015, No. 51. ISAAA:Ithaca, NY
[2] 黄大昉.我国转基因作物育种发展回顾与思考[J].生物工程杂志,2015,31(6):892-900 HUANG Da-fang. Review of transgenic crop breeding in China [J]. Chinese Journal of Biotechology, 2015, 31(6):892-900
[3] Chhapekar S, Raghavendrarao S, Pavan G, et al. Transgenic rice expressing a codon-modified synthetic CP4-EPSPS confers tolerance to broad-spectrum herbicide, glyphosate [J].Plant Cell Report, 2015, 34(5): 721-731
[4] EFSA GMO Working Panel. Safety and nutritional assessment of GM plants and derived foods: the role of animal feeding trials [J]. Food and Chemical Toxicology,2008, 46: S2-S70
[5] He X, de Brum P A, Chukwudebe A, et al. Rat and poultry feeding studies with soybean meal produced from imidazolinone-tolerant (CV127) soybeans [J]. Food Chemical Toxicology, 2016, 88: 48-56
[6] Haryu Y, Taguchi Y, Itakura E, et al. Longterm biosafety assessment of a genetically modified (GM) plant: the genetically modified (GM) insect-resistant Bt11 corn does not affect the performance of multi- generations or life span of mice [J]. The Open Plant Science Journal, 2009, 3: 49-53
[7] Battistelli S, Citterio B, Baldelli B, et al. Histochemical and morpho-metrical study of mouse intestine epithelium after a long term diet containing genetically modified soybean [J].European Journal of Histochemistry, 2010, 54(3): e36
[8] Buzoianu S G, Walsh M C, Rea M C, et al. Effect of feeding genetically modified Bt MON810 maize to: 40-day-old pigs for 110 days on growth and health indicators [J]. Animal,2012, 6(10): 1609-1619
[9] 刘金.转 Bar基因抗除草剂稻谷对小鼠的安全性评价[D].长沙:湖南师范大学博士论文,2012 LIU Jin. Safety assessment of bar-transgenic rice with herbicide resistance using a mouse (Mus musculus) model[D]. Changsha: Hunan Normal University, 2012
[10] 富莉娜,颜亨梅,段妍慧,等.转 Bar基因抗除草剂稻谷对妊娠小鼠亚慢性毒性的响应[J].安徽农业科学,2012,40(11):6570-6572 FU Li-na, YAN Heng-mei, DUAN Yan-hui, et al. Effects of the bar-gene genetically modified rice on subchronic toxicity of pregnancy mouse [J]. Journal of Anhui Agricultural Sciences, 2012, 40(11): 6570-6572
[11] 段妍慧,富丽娜,黄毅,等.转 Bar基因稻谷对小鼠肝功能若干参数的影响[J].湖南农业科学,2012,11:4-7 DUAN Yan-hui, FU Li-na, HUANG Yi, et al. Effects of bar-transgenic rice on the parameters of liver function of mice[J]. Hunan Agricultural Sciences, 2012, 11: 4-7
[12] Velimirov A, Binter C, Zentek J. Biological effects of transgenic maize NK603×MON810 fed in long term reproduction studies in mice [J]. Report, Federal Ministry of Health, Family and Youth, 2008
[13] 张珍誉,刘立军,张琳,等.转Bt基因稻谷对小鼠的亚慢性毒性实验[J].毒理学杂志,2010,24(4):126-129 ZHANG Zhen-yu, LIU Li-jun, ZHANG Lin, et al. The subchronic toxicity of the bar-gene genetically modified on rice [J]. Journal of Toxicology, 2010, 24(4): 126-129
[14] Tudisco R, Mastellone V, Cutrignelli M I, et al. Fate of transgenic DNA and evaluation of metabolic effects in goats fed genetically modified soybean and in their offsprings [J].Animal, 2010, 4(10): 1662-1671
[15] Séralini G E, Clair E, Mesnage R, et al. Republished study:Long term toxicity of a Roundup herbicide and a Roundup-tolerant genetically modified maize [J].Environmental Sciences Europe, 2014, 26(1): 14
[16] Mesnage R, Arno M, Séralini G E, et al. Transcriptome and metabolome analysis of liver and kidneys of rats chronically fed NK603 roundup-tolerant genetically modified maize [J].Environmental Science Europe, 2017, 29(1): 6
[17] Han F, Zhou D, Liu X, et al. Attitudes in China about crops and foods developed by biotechnology [J]. PLoS One, 2015,10(9): e0139114
[18] 曲瑛德,陈源泉,侯云鹏,等.我国转基因生物安全调查 I:公众对转基因生物安全与风险的认知[J].中国农业大学学报,2011,16(6):1-10 QU Ying-de, CHEN Yuan-quan, HOU Yun-peng, et al.Survey analysis of the cognition of GMO risk and safety among Chinese public [J]. Journal of China Agricultural University, 2011, 16(6): 1-10
[19] Wang M, Jiang S, Wang Y F. Recent advances in the production of recombinant subunit vaccines in Pichia pastoris [J]. Bioengineered, 2016, 7(3): 155-165
[20] Wagner J M, Apler H S. Synthetic biology and molecular genetics in non-conventional yeasts: current tools and future advances [J]. Fungal Genetics and Biology, 2016, 89:126-136
[21] Ergun B G, Calik P. Lignocellulose degrading extremozymes produced by Pichia pastoris: current status and future prospects [J]. Bioprocess and Biosystems Engineering, 2016,39(1): 1-36
[22] Cregg J M, Tolstorukov I, Kusari A, et al. Expression in the yeast Pichia pastoris [J]. Methods in Enzymology, 2009, 463:169-89
[23] 尹春光,杜立新,赵桂平,等.Mx基因稀有密码子和mRNA结构及大肠杆菌表达优化[J].遗传,2009,31(1):75-82 YIN Chun-guang, DU Li-xin, ZHAO Gui-ping, et al.Optimizing the expression of Mx gene in Escherichia coli based on rare codon and mRNA structure [J]. Hereditas, 2009,31(1): 75-82
[24] Presnyak V, Alhusaini N, Chen Y H, et al. Codon optimality is a major determinant of mRNA stability [J]. Cell, 2015,160(6): 1111-1124
[25] Kudla G, Lipinski L, Caffin F, et al. High guanine and cytosine content increases mRNA levels in mammalian cells[J]. PLoS Biology, 2006, 4(6): e180
[26] Newman Z R, Young J M, Ingolia N T, et al. Differences in codon bias and GC content contribute to the balanced expression of TLR7 and TLR9 [J]. PNAS, 2016, 113(10):E1362-1371
[27] 翁绿水,陈芬,肖国樱,等.Cry2Aa基因优化设计及功能验证[J].农业生物技术学报,2013,21(11):1261-1269 WENG Lv-shui, CHEN Fen, XIAO Guo-ying, et al.Optimization and Functional verification of Cry2Aa gene [J].Journal of Agricultural Biotechnology, 2013, 21(11):1261-1269
[28] 周宗梁,林智敏,耿丽丽,等.水稻中 cry1Ah1基因密码子优化方案的比较[J].生物工程学报,2012,28(10):1184-1194 ZHOU Zong-liang, LIN Zhi-min, GENG Li-li, et al.Comparison of codon optimizations of cry1Ah1 gene in rice[J]. Chinese Journal of Biotechnology, 2012, 28(10):1184-1194
[29] Kozak M. An analysis of vertebrate mRNA sequences:intimations of translational control [J]. The Journal of Cell Biology, 1991, 115(4): 887-903
[30] Kozak M. Pushing the limits of the scanning mechanism for initiation of translation [J]. Gene, 2002, 299(1-2): 1-34
[31] 刘树鹏,李刚强,王楠,等.CP4-EPSPS蛋白在大肠杆菌中的表达与制备[J].中国农业科技导报,2012,14(1):91-97 LIU Shu-peng, LI Gang-qiang, WANG Nan, et al. Expression in E. coli and preparation of CP4-EPSPS protein [J]. Journal of Agricultural Science and Technology, 2012, 14(1): 91-97
[32] 柴帅杰,武鹏程,荣瑞娟,等.转基因水稻中CP4-EPSPS蛋白质的检测及其表达特征研究[J].核农学报,2017,31(1):44-50 CHAI Shuai-jie, WU Peng-cheng, RONG Rui-juan, et al.Detection and expression characteristics of CP4-EPSPS protein in transgenic rice [J]. Journal of Nuclear Agricultural Sciences, 2017, 31(1): 44-50
附件1
cp4-epsps基因优化序列:
ATGTTGCATGGTGCCTCCTCCAGACCTGCTACTGCTCGTAAGTCTTCCGGATTGTCTGGAACCGTCCGT ATCCCAGGTGATAAGTCCATCTCCCACCGTTCCTTCATGTTCGGTGGTTTGGCCTCTGGAGAGACCAGAAT TACCGGTTTGTTGGAAGGTGAAGACGTCATCAACACCGGTAAGGCTATGCAGGCTATGGGTGCTAGAATC CGTAAGGAAGGTGACACCTGGATTATCGACGGAGTCGGAAACGGTGGTCTTTTGGCTCCTGAGGCCCCTTT GGATTTTGGAAACGCTGCCACCGGTTGCAGACTTACCATGGGTCTTGTCGGAGTCTACGACTTCGACTCCA CCTTCATTGGTGACGCTTCCTTGACCAAGCGTCCAATGGGTAGAGTCTTGAACCCATTGCGTGAGATGGGT GTTCAGGTTAAGTCTGAGGACGGTGACAGATTGCCTGTCACCTTGAGAGGTCCTAAGACCCCAACCCCAAT TACCTACCGTGTCCCTATGGCCTCTGCTCAAGTTAAGTCTGCCGTCTTGTTGGCCGGTTTGAACACTCCAGG AATCACCACTGTCATCGAGCCTATTATGACTTGCGACCACACCGAGAAAATGTTGCAGGGTTTCGGTGCTA ACTTGACCGTTGAAACCGACGCCGACGGAGTTAGAACTATTAGATTGGAGGGTAGAGGTAAATTGACTGG TCAGGTCATCGATGTTCCAGGAGATCCATCCTCCACTGCTTTCCCACTTGTCGCTGCTTTGTTGGTCCCAGG TTCCGACGTTACCATTTTGAACGTTTTGATGAATCCAACCAGAACCGGATTGATTTTGACTTTGCAAGAAA TGGGTGCCGACATCGAGGTCATCAACTTGAGATTGGCCGGTGGTGAGGATGTCGCTGATCTTCGTGTCCGT TCCTCTACCTTGAAGGGTGTTACCGTCCCAGAGGATCGTGCCCCTCCAATGATCGACGAGTACCCAATCTT GGCTGTCGCTGCTGCTTTCGCTGAAGGTGCCACTGTTATGAACGGATTGGAGGAGTTGCGTGTCAAGGAAT CTGACAGATTGTCTGCCGTCGCCAACGGATTGAAGTTGAACGGTGTCGATTGCGACGAGGGTGAGACTTCT TTGGTCGTTAGAGGTCGTCCAGACGGTAAGGGTTTGGGTAACGCTTCTGGTGCTGCTGTTGCTACCCACTT GGACCACAGAATCGCCATGTCCTTCTTGGTCATGGGATTGGTCTCCGAAAACCCAGTCACCGTCGACGACG CTACCATGATTGCTACTTCCTTCCCTGAGTTTATGGACTTGATGGCCGGTCTTGGTGCTAAGATCGAGTTGT CCGATACCAAGGCTGCT
CP4-EPSPS氨基酸序列:
MLHGASSRPATARKSSGLSGTVRIPGDKSISHRSFMFGGLASGETRITGLLEGEDVINTGKAMQAMGARIR KEGDTWIIDGVGNGGLLAPEAPLDFGNAATGCRLTMGLVGVYDFDSTFIGDASLTKRPMGRVLNPLREMGVQ VKSEDGDRLPVTLRGPKTPTPITYRVPMASAQVKSAVLLAGLNTPGITTVIEPIMTCDHTEKMLQGFGANLTVE TDADGVRTIRLEGRGKLTGQVIDVPGDPSSTAFPLVAALLVPGSDVTILNVLMNPTRTGLILTLQEMGADIEVIN LRLAGGEDVADLRVRSSTLKGVTVPEDRAPPMIDEYPILAVAAAFAEGATVMNGLEELRVKESDRLSAVANGL KLNGVDCDEGETSLVVRGRPDGKGLGNASGAAVATHLDHRIAMSFLVMGLVSENPVTVDDATMIATSFPEFM DLMAGLGAKIELSDTKAA
Expression and Identification of CP4-EPSPS Gene in Pichia pastoris
Abstract: Constructing the genetically modified (GM) Pichia pastoris stains is the key step forestablishing a new method to assess the safety of foreign protein . Based on the research model of the 5-enolpyruvyl shikinmate-3-phosphate synthase derived from Agrobacterium CP4 strain (CP4-EPSPS), the cp4-epsps gene was chemically synthesized after optimizing thecodon usage bias. The codon adaption index (CAI) of optimized sequence was 0.85, and the GC content of optimized sequence was 52%. Cp4-epsps gene was amplified by PCR and cloned to the expression vector pPICZb of Pichia pastoris GS115 through homologous end-joining, and the recombinant vector pPICZb-EPSPS was constructed. The correct recombinant vector was transferred into GS115 strain through electroporation, and four positive strains were screened through the right site via PCR. Then, those positive strains were induced by 0.5% methanol for four days at 28 ℃, 250~300 r/min. Finally, the expression of cp4-epsps in GS115 was identified from CP4-EPSPS monoclonal antibody and C-MYC monoclonal antibody by SDS-PAGE and western blotting. The results showed that the target gene was expressed in GS115 strain and MW of protein was 50 ku, which coincided with the theoretical results. In conclusion, the construction of transgenic Pichia pastoris GS115 strains is beneficial for the safety assessment of transgenic CP4-EPSPS synthase in GM crops.
Key words: Cp4-epsps gene; Pichia pastoris; transgenic ingredients; safety assessment
文章篇号:1673-9078(2017)11-55-62
DOI:10.13982/j.mfst.1673-9078.2017.11.009
收稿日期:2017-03-15
基金项目:环保部公益性行业科研专项(201509044)